Piruvato deshidroxenase
- Ver tamén complexo da piruvato deshidroxenase. Non confundir con piruvato descarboxilase.
| piruvato deshidroxenase (transferente de acetilo) | |||||||||
|---|---|---|---|---|---|---|---|---|---|

| |||||||||
| Estrutura cristalográfica da piruvato deshidroxenase. É un dímero de seis dominios con rexións α (azul), α’ (anarela), β (vermella), e β’ (verde azulada) indicadas en distintas cores. O pirofosfato de tiamina (TPP) móstrase como en gris, dous ións magnesio en púrpura en ligamento metálico co TPP, e dous ións potasio en laranxa.[1][2] | |||||||||
| Identificadores | |||||||||
| Número EC | 1.2.4.1 | ||||||||
| Número CAS | 9014-20-4 | ||||||||
| Bases de datos | |||||||||
| IntEnz | vista de IntEnz | ||||||||
| BRENDA | entrada de BRENDA | ||||||||
| ExPASy | vista de NiceZyme | ||||||||
| KEGG | entrada de KEGG | ||||||||
| MetaCyc | vía metabólica | ||||||||
| PRIAM | perfil | ||||||||
| Estruturas PDB | RCSB PDB PDBe PDBj PDBsum | ||||||||
| Gene Ontology | AmiGO / EGO | ||||||||
| |||||||||
A piruvato deshidroxenase (E1) é o primeiro encima compoñente do complexo da piruvato deshidroxenase (PDC). O complexo da piruvato deshidroxenase transforma o piruvato en acetil-CoA por medio dun proceso chamado descarboxilación do piruvato, e o seu compoñente a piruvato deshidroxenase cataliza a primeira das reaccións do proceso. Este acetil-CoA pode despois incorporarse ao ciclo do ácido cítrico para levar a cabo a respiración celular, polo que este encima contribúe a conectar a glicólise co ciclo do ácido cítrico.
Función
[editar | editar a fonte]A piruvato deshidroxenase (E1) realiza a primeira das reaccións que teñen lugar no complexo da piruvato deshidroxenase (PDC), que é unha descarboxilación do substrato 1 (piruvato) e unha acetilación redutiva do substrato 2 (ácido lipoico). O ácido lipoico está unido covalentemente á dihidrolipoamida acetiltransferase (E2), que é o segundo encima catalítico compoñente do complexo da piruvato deshidroxenase, que recolle os produtos de E1 para transformalos. A reacción catalizada pola piruvato deshidroxenase (E1) considérase que é o paso limitante para o complexo da piruvato deshidroxenase (PDC).
Regulación
[editar | editar a fonte]A fosforilación de E1 polo encima piruvato deshidroxenase quinase (PDK) inactiva a E1 e, en consecuencia, a todo o complexo. Esta acción é revertida pola piruvato deshidroxenase fosfatase. A piruvato deshidroxenase fosfatase é estimulada pola insulina, o fosfoenolpiruvato (PEP), e o AMP, pero é inhibida competitivamente polo ATP, NADH, e acetil-CoA.

Mecanismo
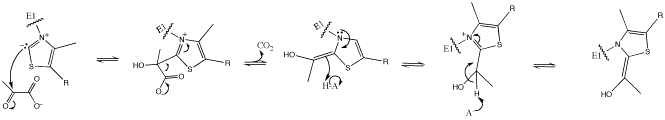
[editar | editar a fonte]Na reacción intervén o coencima pirofosfato de tiamina (TPP) unido ao encima. A forma de resonancia iluro do pirofosfato de tiamina comeza atacando o grupo cetona electrofílico do piruvato. Despois, o intermediato β-alcóxido descarboxílase e o enol resultante é desprotonado no átomo de carbono para formar un dipolo 1,3 estabilizado no que está implicado un átomo de nitróxeno cargado positivamente do heterociclo da tiamina. Este dipolo 1,3 sofre unha acetilación redutiva coa lipoamida-E2.
Os datos bioquímicos e estruturais que se teñen de E1 indican un mecanismo de activación do cofactor TPP por formación dun enlace de hidróxeno conservado co residuo de glutamato (Glu59 na E1 humana) e pola imposición dunha conformación V que fai que o átomo N4’ da aminopirimidina estableza un enlace de hidróxeno intramolecular co átomo C2 do tiazolio. Esta combinación única de contactos e conformacións da TPP orixina finalmente a formación do carbanión C2 reactivo. Unha vez que o cofactor TPP descarboxila o piruvato, a porción acetilo convértese nun derivado hidroxietil unido covalentemente ao TPP.[1]
Estrutura
[editar | editar a fonte]E1 é unha proteína multimérica. As E1s demamíferos, incluíndo os humanos, son tetraméricas, e están compostas por dúas subunidades α e dúas β.[1] Algunhas E1s bacterianas, incluída a E1 de Escherichia coli, están compostas por dúas subunidades similares, cada unha das cales é tan grande coma a suma das masas moleculares das subunidades α e β.[3]

Sitio activo
[editar | editar a fonte]
E1 ten dous sitios catalíticos, cada un deles cos cofactores pirofosfato de tiamina (TPP) e ión magnesio. A subunidade α únese ao ión magnesio e ao fragamento pirofosfato mentres que a subunidade β se une ao fragmento pirimidina da TPP, formando o conxunto o sitio catalítico na interface das subunidades.[1]
No sitio activo da piruvato deshidroxenase (ver imaxe da esquerda creada a partir de PDB 1NI4) a TPP está unida por ligamento metálico a un ión magnesio (esfera púrpura) e por medio de enlaces de hidróxeno con aminoácidos. Aínda que se poden atopar uns 20 aminoácidos no sitio activo, os aminoácidos Tyr 89, Arg 90, Gly 136, Val 138, Asp 167, Gly 168, Ala 169, Asn, 196, e His 263 son os que realmente participan nos enlaces de hidróxeno que sosteñen no sitio activo ao TPP e ao piruvato (non mostrado aquí). Os aminoácidos móstranse como arames, e o TPP como bólas e barras. O sitio activo tamén axuda á transferencia do acilo da TPP á lipoamida situada en E2, que está agardando pola seguinte reacción.[1][2]
Patoloxía
[editar | editar a fonte]A piruvato deshidroxenase é un autoantíxeno recoñecido autoinmunitariamente polo sistema inmunitario na cirrose biliar primaria. Os autoanticorpos que se forman parecen recoñecer a proteína oxidada que se orixina en respostas inmunes inflamatorias. Algunhas destas respostas inflamatorias poderían estar relacionadas coa sensibilidade ao glute xa que arredor do 50% dos pacientes de insuficiencia hepática aguda nun estudo presentaban un autoanticorpo non mitocondrial contra a transglutaminase tisular.[4] Outros autoantíxenos mitocondriais son a oxoglutarato deshidroxenase e o complexo da alfa-cetoácido de cadea ramificada deshidroxenase, que son antíxenos recoñecidos polos anticorpos anitimitocondriais.
A deficiencia de piruvato deshidroxenase é unha enfermidade conxénita metabólica dexenerativa que se orixina por unha mutación do complexo da piruvato deshidroxenase (PDC) localizado no cromosoma X. Aínda que se identificaron defectos en todos os encimas do complexo, o compoñente E1-α é predominantemente o culpable da doenza. O mal funcionamento do ciclo do ácido cítrico debido á deficiencia de piruvato deshidroxenase priva ao corpo de enerxía e causa unha acumulación anormal de lactato. A deficiencia de piruvato deshidroxenase é unha causa común de acidose láctica en neonatos e a miúdo preséntase con fatiga grave, mala alimentación, hiperventilación (taquipnea), e nalgúns casos morte.[5]
Exemplos
[editar | editar a fonte]Entre as proteínas humanas con actividade de piruvato deshidroxenase están:
|
|
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Encimas relacionados
[editar | editar a fonte]Nas bacterias, existe unha forma de piruvato deshidroxenase (tamén chamada piruvato oxidase, EC 1.2.2.2), que conecta a oxidación do piruvato a acetato e dióxido de carbono coa redución de ferrocitocromo. En E. coli este encima está codificado polo xene pox B e a proteína ten un cofactor flavínico.[6] Este encima incrementa a eficacia do crecemento de E. coli en condicións aeróbicas.[7]
Notas
[editar | editar a fonte]- ↑ 1,0 1,1 1,2 1,3 1,4 PDB 1ni4; Ciszak EM, Korotchkina LG, Dominiak PM, Sidhu S, Patel MS (2003). "Structural basis for flip-flop action of thiamin pyrophosphate-dependent enzymes revealed by human pyruvate dehydrogenase". J. Biol. Chem. 278 (23): 21240–6. PMID 12651851. doi:10.1074/jbc.M300339200.
- ↑ 2,0 2,1 Molecular graphics images were produced using the UCSF Chimera package from the Resource for Biocomputing, Visualization, and Informatics at the University of California, San Francisco; Pettersen EF, Goddard TD, Huang CC, Couch GS, Greenblatt DM, Meng EC, Ferrin TE (2004). "UCSF Chimera—a visualization system for exploratory research and analysis". J Comput Chem 25 (13): 1605–12. PMID 15264254. doi:10.1002/jcc.20084.
- ↑ Arjunan P, Nemeria N, Brunskill A, Chandrasekhar K, Sax M, Yan Y, Jordan F, Guest JR, Furey W. (2002). "Structure of the pyruvate dehydrogenase multienzyme complex E1 component from Escherichia coli at 1.85 A resolution". Biochemistry 41 (16): 5213–21. PMID 11955070. doi:10.1021/bi0118557.
- ↑ Leung PS, Rossaro L, Davis PA; et al. (2007). "Antimitochondrial antibodies in acute liver failure: Implications for primary biliary cirrhosis". Hepatology 46 (5): 1436. PMID 17657817. doi:10.1002/hep.21828.
- ↑ Frye, Richard E., and Paul J. Benke. "Pyruvate Dehydrogenase Complex Deficiency." EMedicine. 11 Dec. 2007. WebMD. 14 Dec. 2008 <http://emedicine.medscape.com/article/948360-overview>.
- ↑ Recny MA, Hager LP (1982). "Reconstitution of native Escherichia coli pyruvate oxidase from apoenzyme monomers and FAD". J. Biol. Chem. 257 (21): 12878–86. PMID 6752142.
- ↑ Abdel-Hamid AM, Attwood MM, Guest JR (2001). "Pyruvate oxidase contributes to the aerobic growth efficiency of Escherichia coli". Microbiology (Reading, Engl.) 147 (Pt 6): 1483–98. PMID 11390679.
Véxase tamén
[editar | editar a fonte]Outros artigos
[editar | editar a fonte]- Descarboxilación do piruvato
- Complexo da piruvato deshidroxenase (formado por varias unidades encimáticas, entre elas a piruvato deshidroxenase)
- Piruvato descarboxilase (un encima completamente diferente que forma acetaldehido e intervén na fermentación alcohólica)
Ligazóns externas
[editar | editar a fonte]- Pyruvate Dehydrogenase-E1 Medical Subject Headings (MeSH) na Biblioteca Nacional de Medicina dos EUA.
- www.brookscole.com Animacións Arquivado 24 de xullo de 2012 en Wayback Machine.