mTOR
| mechanistic target of rapamycin kinase | |||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||||||||||||||||||||||||||||||||||||||||||||
| Nomes alternativos | |||||||||||||||||||||||||||||||||||||||||||||||||||
| IDs externos | GeneCards: [1] | ||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Wikidata | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
O alvo da rapamicina em mamíferos (mammalian target of rapamycin - mTOR),[1] também chamado de alvo mecanístico da rapamicina (mechanistic target of rapamycin) e, às vezes, de proteína 12 de ligação FK506 (FK506-binding protein 12-rapamycin-associated protein 1 - FRAP1), é uma quinase que, em humanos, é codificada pelo gene MTOR.[2][3][4] O mTOR é um membro da família de proteínas quinases relacionadas à fosfatidilinositol 3-quinase.[5]
O mTOR se liga a outras proteínas e atua como um componente central de dois complexos proteicos distintos, o complexo mTOR 1 e o complexo mTOR 2, que regulam diferentes processos celulares.[6] Em particular, como um componente central de ambos os complexos, o mTOR funciona como uma proteína quinase serina/treonina que regula o crescimento celular, a proliferação celular, a motilidade celular, a sobrevivência celular, a síntese proteica, a autofagia e a transcrição.[6][7] Como componente central do mTORC2, o mTOR também funciona como uma proteína quinase de tirosina que promove a ativação dos receptores de insulina e dos receptores do fator de crescimento semelhante à insulina tipo 1.[8] O mTORC2 também foi implicado no controle e na manutenção do citoesqueleto de actina.[6][9]
Descoberta
[editar | editar código-fonte]Rapa Nui (Ilha de Páscoa - Chile)
[editar | editar código-fonte]O estudo do TOR teve origem na década de 1960 com uma expedição à Ilha de Páscoa (conhecida pelos habitantes da ilha como Rapa Nui), com o objetivo de identificar produtos naturais de plantas e do solo com possível potencial terapêutico. Em 1972, Suren Sehgal identificou uma pequena molécula de uma bactéria do solo, Streptomyces hygroscopicus, que ele purificou e inicialmente relatou possuir uma potente atividade antifúngica. Ele a batizou apropriadamente de rapamicina, observando sua fonte e atividade originais.[10][11] No entanto, os primeiros testes revelaram que a rapamicina também tinha uma potente atividade imunossupressora e citostática contra o câncer. A rapamicina não recebeu inicialmente um interesse significativo do setor farmacêutico até a década de 1980, quando a Wyeth-Ayerst apoiou os esforços de Sehgal para investigar mais a fundo o efeito da rapamicina no sistema imunológico. Isso acabou levando à sua aprovação pela FDA como imunossupressor após o transplante renal. Entretanto, antes de sua aprovação pela FDA, o funcionamento da rapamicina permanecia completamente desconhecido.
Histórico subsequente
[editar | editar código-fonte]A descoberta do TOR e do mTOR foi resultado de estudos independentes do produto natural rapamicina por Joseph Heitman, Rao Movva e Michael N. Hall em 1991;[12] por David M. Sabatini, Hediye Erdjument-Bromage, Mary Lui, Paul Tempst e Solomon H. Snyder[3] em 1994; e por Candace J. Sabers, Mary M. Martin, Gregory J. Brunn, Josie M. Williams, Francis J. Dumont, Gregory Wiederrecht e Robert T. Abraham em 1995.[4] Em 1991, trabalhando com levedura, Hall e seus colegas identificaram os genes TOR1 e TOR2.[12] Em 1993, Robert Cafferkey, George Livi e seus colegas, e Jeannette Kunz, Michael N. Hall e seus colegas clonaram independentemente os genes que mediam a toxicidade da rapamicina em fungos, conhecidos como genes TOR/DRR.[13][14] Entretanto, o alvo molecular do complexo FKBP12-rapamicina em mamíferos não era conhecido. Em 1994, pesquisadores que trabalhavam nos laboratórios de Stuart L. Schreiber, Solomon H. Snyder e Robert T. Abraham descobriram independentemente uma proteína que interage diretamente com a FKBP12-rapamicina, que ficou conhecida como mTOR devido à sua homologia com os genes TOR/DRR de levedura.[2][3][4]
A rapamicina interrompe a atividade fúngica na fase G1 do ciclo celular. Em mamíferos, ela suprime o sistema imunológico bloqueando a transição da fase G1 para S nos linfócitos T.[15] Assim, ela é usada como imunossupressor após o transplante de órgãos.[16] O interesse na rapamicina foi renovado após a descoberta do produto natural imunossupressor estruturalmente relacionado FK506 em 1987. Em 1989-90, foi determinado que o FK506 e a rapamicina inibiam as vias de sinalização do receptor de células T (TCR) e do receptor de IL-2, respectivamente.[17][18] Os dois produtos naturais foram usados para descobrir as proteínas de ligação do FK506 e da rapamicina, incluindo a FKBP12, e para fornecer evidências de que a FKBP12-FK506 e a FKBP12-rapamicina poderiam agir por meio de mecanismos de ganho de função que visam funções celulares distintas. Essas investigações incluíram estudos importantes de Francis Dumont e Nolan Sigal na Merck, que contribuíram para mostrar que o FK506 e a rapamicina se comportam como antagonistas recíprocos.[19][20] Esses estudos implicaram o FKBP12 como um possível alvo da rapamicina, mas sugeriram que o complexo poderia interagir com outro elemento da cascata mecanicista.[21][22]
Em 1991, a calcineurina foi identificada como alvo da FKBP12-FK506.[23] O alvo da FKBP12-rapamicina permaneceu misterioso até que estudos genéticos e moleculares em leveduras estabeleceram a FKBP12 como alvo da rapamicina e implicaram TOR1 e TOR2 como alvos da FKBP12-rapamicina em 1991 e 1993,[12][24] seguidos por estudos em 1994, quando vários grupos, trabalhando independentemente, descobriram a quinase mTOR como seu alvo direto em tecidos de mamíferos.[2][3][16] A análise da sequência do mTOR revelou que ela é o ortólogo direto das proteínas codificadas pelos genes TOR1 e TOR2 (yeast target of rapamycin 1 e 2), que Joseph Heitman, Rao Movva e Michael N. Hall identificaram em agosto de 1991 e maio de 1993. Independentemente, George Livi e colegas relataram posteriormente os mesmos genes, que chamaram de resistência dominante à rapamicina 1 e 2 (DRR1 e DRR2), em estudos publicados em outubro de 1993.
A proteína, agora chamada de mTOR, foi originalmente denominada FRAP por Stuart L. Schreiber e RAFT1 por David M. Sabatini;[2][3] FRAP1 foi usado como seu símbolo gênico oficial em humanos. Devido a esses nomes diferentes, mTOR, que foi usado pela primeira vez por Robert T. Abraham,[2] foi cada vez mais adotado pela comunidade de cientistas que trabalham na via mTOR para se referir à proteína e em homenagem à descoberta original da proteína TOR em levedura que foi denominada TOR, o Alvo da Rapamicina (Target of Rapamycin), por Joe Heitman, Rao Movva e Mike Hall. A TOR foi originalmente descoberta no Biozentrum e na Sandoz Pharmaceuticals em 1991, na Basileia, Suíça, e o nome TOR é uma homenagem a essa descoberta, já que tor significa porta ou portão em alemão, e a cidade da Basileia já foi cercada por uma muralha pontuada por portões para a cidade, incluindo o icônico Spalentor.[25] Inicialmente, "mTOR" significava "mammalian target of rapamycin" (alvo mamífero da rapamicina), mas o significado do "m" foi posteriormente alterado para "mechanistic" (mecanicista).[26] Da mesma forma, com as descobertas subsequentes, o TOR do peixe-zebra foi denominado zTOR, o TOR da Arabidopsis thaliana foi denominado AtTOR e o TOR da Drosophila foi denominado dTOR. Em 2009, o nome do gene FRAP1 foi oficialmente alterado pelo Comite de Nomenclatura de Genes (HGNC) da Organização Genoma Humano (HUGO) para mTOR, que significa mechanistic target of rapamycin (alvo mecânico da rapamicina).[27]
A descoberta do TOR e a subsequente identificação do mTOR abriram as portas para o estudo molecular e fisiológico do que hoje é chamado de via mTOR e teve um efeito catalisador no crescimento do campo da biologia química, em que pequenas moléculas são usadas como sondas da biologia.
Função
[editar | editar código-fonte]O mTOR integra a entrada das vias ascendentes, incluindo insulina, fatores de crescimento (como IGF-1 e IGF-2) e aminoácidos.[7] O mTOR também detecta os níveis celulares de nutrientes, oxigênio e energia.[28] A via mTOR é uma reguladora central do metabolismo e da fisiologia dos mamíferos, com papéis importantes na função dos tecidos, incluindo fígado, músculo, tecido adiposo branco e marrom,[29] e o cérebro, e é desregulada em doenças humanas, como diabetes, obesidade, depressão e certos tipos de câncer.[30][31] A rapamicina inibe o mTOR associando-se ao seu receptor intracelular FKBP12.[32][33] O complexo FKBP12-rapamicina liga-se diretamente ao domínio de ligação FKBP12-Rapamicina (FRB) do mTOR, inibindo sua atividade.[33]
Nas plantas
[editar | editar código-fonte]As plantas expressam o alvo mecanicista da rapamicina (mTOR) e têm um complexo de quinase TOR. Nas plantas, apenas o complexo TORC1 está presente, ao contrário do alvo de rapamicina dos mamíferos, que também contém o complexo TORC2.[34] As espécies de plantas têm proteínas TOR nos domínios de ligação à proteína quinase e à FKBP-rapamicina (FRB) que compartilham uma sequência de aminoácidos semelhante à do mTOR em mamíferos.[35]
Papel do mTOR nas plantas
[editar | editar código-fonte]O complexo TOR kinase é conhecido por ter uma função no metabolismo das plantas. O complexo TORC1 é ativado quando as plantas estão vivendo as condições ambientais adequadas para sobreviver. Uma vez ativadas, as células vegetais passam por reações anabólicas específicas. Essas reações incluem o desenvolvimento da planta, a tradução do mRNA e o crescimento das células dentro da planta. No entanto, a ativação do complexo TORC1 impede a ocorrência de processos catabólicos, como a autofagia.[34] Descobriu-se que a sinalização da quinase TOR nas plantas ajuda na senescência, na floração, no crescimento da raiz e da folha, na embriogênese e na ativação do meristema acima da coifa da raiz de uma planta.[36] Descobriu-se também que o mTOR está altamente envolvida no desenvolvimento do tecido embrionário das plantas.[35]
Complexos
[editar | editar código-fonte]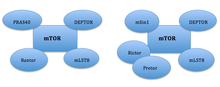
O mTOR é a subunidade catalítica de dois complexos estruturalmente distintos: mTORC1 e mTORC2.[37] Os dois complexos se localizam em diferentes compartimentos subcelulares, afetando assim sua ativação e função.[38] Após a ativação por Rheb, o mTORC1 se localiza no complexo Ragulator-Rag na superfície do lisossomo, onde se torna ativa na presença de aminoácidos suficientes.[39][40]
mTORC1
[editar | editar código-fonte]O Complexo mTOR 1 (mTORC1) é composto por mTOR, proteína associada à regulação de mTOR (Raptor), proteína 8 letal de mamífero com SEC13 (mLST8) e os componentes não essenciais PRAS40 e DEPTOR.[41][42] Esse complexo funciona como um sensor de nutrientes/energia/redox e controla a síntese de proteínas.[7][41] A atividade do mTORC1 é regulada pela rapamicina, insulina, fatores de crescimento, ácido fosfatídico, determinados aminoácidos e seus derivados (por exemplo, l-leucina e β- e ácido β-hidroxi β-metilbutírico), estímulos mecânicos e estresse oxidativo.[41][43][44]
mTORC2
[editar | editar código-fonte]O Complexo mTOR 2 (mTORC2) é composto por mTOR, proteína companheira de mTOR insensível à rapamicina (RICTOR), mLST8 e proteína de interação a proteína quinase ativada por estresse de mamíferos (mSIN1).[45][46] Foi demonstrado que o mTORC2 funciona como um importante regulador do citoesqueleto de actina por meio de sua estimulação das fibras de estresse de F-actina, paxilina, RhoA, Rac1, Cdc42 e proteína quinase C α (PKCα).[46] O mTORC2 também fosforila a proteína quinase serina/treonina Akt/PKB no resíduo de serina Ser473, afetando assim o metabolismo e a sobrevivência.[47] A fosforilação do resíduo de serina Ser473 da Akt pelo mTORC2 estimula a fosforilação da Akt no resíduo de treonina Thr308 pelo PDK1 e leva à ativação total da Akt.[48][49] Além disso, o mTORC2 apresenta atividade de proteína quinase de tirosina e fosforila o receptor do fator de crescimento semelhante à insulina tipo 1 (IGF-1R) e o receptor de insulina (InsR) nos resíduos de tirosina Tyr1131/1136 e Tyr1146/1151, respectivamente, levando à ativação total do IGF-IR e do InsR.[8]
Inibição pela rapamicina
[editar | editar código-fonte]A rapamicina (Sirolimus) inibe o mTORC1, resultando na supressão da senescência celular,[50] o que parece proporcionar a maioria dos efeitos benéficos do medicamento (incluindo a extensão do tempo de vida em estudos com animais). A supressão da resistência à insulina pelas sirtuínas é responsável por pelo menos parte desse efeito.[51] A sirtuína 3 prejudicada leva à disfunção mitocondrial.[52]
A rapamicina tem um efeito mais complexo sobre o mTORC2, inibindo-o somente em determinados tipos de células sob exposição prolongada. A interrupção do mTORC2 produz sintomas semelhantes aos do diabetes, como diminuição da tolerância à glicose e insensibilidade à insulina.[53]
Experimentos com deleção gênica
[editar | editar código-fonte]A via de sinalização mTORC2 é menos definida do que a via de sinalização mTORC1. As funções dos componentes dos complexos mTORC foram estudadas com o uso de knockdowns e knockouts de genes e foram encontrados os seguintes fenótipos:
- NIP7: O knockdown reduziu a atividade de mTORC2, o que é indicado pela diminuição da fosforilação de substratos de mTORC2.[54]
- RICTOR: A superexpressão leva à metástase e o knockdown inibe a fosforilação de PKC induzida pelo fator de crescimento.[55] A deleção constitutiva de Rictor em camundongos leva à letalidade embrionária,[56] enquanto a deleção específica do tecido leva a uma variedade de fenótipos; um fenótipo comum da deleção de Rictor no fígado, no tecido adiposo branco e nas células beta pancreáticas é a intolerância sistêmica à glicose e a resistência à insulina em um ou mais tecidos.[53][57][58][59] A diminuição da expressão de Rictor em camundongos diminui a vida útil dos machos, mas não das fêmeas.[60]
- mTOR: a inibição de mTORC1 e mTORC2 por PP242 [2-(4-Amino-1-isopropil-1H-pirazolo[3,4-d]pirimidin-3-il)-1H-indol-5-ol] leva à autofagia ou apoptose; a inibição de mTORC2 isolada por PP242 evita a fosforilação do sítio Ser-473 em AKT e prende as células na fase G1 do ciclo celular.[61] A redução genética da expressão de mTOR em camundongos aumenta significativamente o tempo de vida.[62]
- PDK1: o knockout é letal; o alelo hipomórfico resulta em menor volume de órgãos e tamanho do organismo, mas ativação normal de AKT.[63]
- AKT: Os camundongos nocauteados apresentam apoptose espontânea (AKT1), diabetes grave (AKT2), cérebros pequenos (AKT3) e deficiência de crescimento (AKT1/AKT2).[64] Os camundongos heterozigotos para AKT1 têm maior tempo de vida.[65]
- TOR1, o ortólogo de S. cerevisiae do mTORC1, é um regulador do metabolismo do carbono e do nitrogênio; as cepas TOR1 KO regulam a resposta ao nitrogênio, bem como a disponibilidade de carbono, indicando que é um transdutor nutricional fundamental na levedura.[66][67]
Significância clínica
[editar | editar código-fonte]Envelhecimento
[editar | editar código-fonte]
Descobriu-se que a diminuição da atividade de TOR aumenta o tempo de vida em S. cerevisiae, C. elegans e D. melanogaster.[68][69][70][71] Confirmou-se que o inibidor de mTOR rapamicina aumenta o tempo de vida em camundongos.[72][73][74][75][76]
Existe a hipótese de que alguns regimes dietéticos, como a restrição calórica e a restrição de metionina, causam o prolongamento da vida útil ao diminuir a atividade do mTOR.[68][69] Alguns estudos sugeriram que a sinalização do mTOR pode aumentar durante o envelhecimento, pelo menos em tecidos específicos como o tecido adiposo, e a rapamicina pode agir em parte bloqueando esse aumento.[77] Uma teoria alternativa é que a sinalização mTOR é um exemplo de pleiotropia antagônica e, embora a sinalização mTOR alta seja boa no início da vida, ela é mantida em um nível inadequadamente alto na velhice. A restrição calórica e a restrição de metionina podem agir, em parte, limitando os níveis de aminoácidos essenciais, incluindo leucina e metionina, que são ativadores potentes de mTOR.[78] Foi demonstrado que a administração de leucina no cérebro de ratos diminui a ingestão de alimentos e o peso corporal por meio da ativação da via mTOR no hipotálamo.[79]
De acordo com a teoria dos radicais livres do envelhecimento,[80] as espécies reativas de oxigênio causam danos às proteínas mitocondriais e diminuem a produção de ATP. Posteriormente, por meio da AMPK sensível ao ATP, a via mTOR é inibida e a síntese de proteínas que consomem ATP é desregulada, pois o mTORC1 inicia uma cascata de fosforilação que ativa o ribossomo.[15] Assim, a proporção de proteínas danificadas é aumentada. Além disso, a interrupção do mTORC1 inibe diretamente a respiração mitocondrial.[81] Esses feedbacks positivos sobre o processo de envelhecimento são neutralizados por mecanismos de proteção: A diminuição da atividade do mTOR (entre outros fatores) regula positivamente a remoção de componentes celulares disfuncionais por meio da autofagia.[80]
O mTOR é um dos principais iniciadores do fenótipo secretor associado à senescência (SASP).[82] A interleucina 1 alfa (IL1A) é encontrada na superfície das células senescentes, onde contribui para a produção de fatores SASP devido a um ciclo de feedback positivo com o NF-κB.[83][84] A tradução do mRNA da IL1A é altamente dependente da atividade do mTOR.[85] A atividade do mTOR aumenta os níveis de IL1A, mediada pela MAPKAPK2.[83] A inibição da mTOR da ZFP36L1 impede que essa proteína degrade as transcrições de vários componentes dos fatores SASP.[86]
Câncer
[editar | editar código-fonte]A ativação excessiva da sinalização mTOR contribui significativamente para o início e o desenvolvimento de tumores, e descobriu-se que a atividade da mTOR está desregulada em muitos tipos de câncer, incluindo carcinomas de mama, próstata, pulmão, bexiga, cérebro e rins e melanomas.[87] Há vários motivos para a ativação constitutiva. Entre os mais comuns estão as mutações no gene supressor de tumor PTEN. A fosfatase PTEN afeta negativamente a sinalização de mTOR ao interferir no efeito de PI3K, um efetor a montante de mTOR. Além disso, a atividade da mTOR é desregulada em muitos cânceres como resultado do aumento da atividade da PI3K ou da Akt.[88] Da mesma forma, a superexpressão dos efetores da mTOR a jusante 4E-BP1, S6K1, S6K2 e eIF4E leva a um prognóstico ruim do câncer.[89] Além disso, as mutações nas proteínas TSC que inibem a atividade da mTOR podem levar a uma condição denominada complexo de esclerose tuberosa, que se apresenta como lesões benignas e aumenta o risco de carcinoma de células renais.[90]
Foi demonstrado que o aumento da atividade do mTOR impulsiona a progressão do ciclo celular e aumenta a proliferação celular, principalmente devido ao seu efeito na síntese de proteínas. Além disso, o mTOR ativo apoia o crescimento do tumor também indiretamente, inibindo a autofagia.[91] O mTOR ativado constitutivamente funciona no fornecimento de oxigênio e nutrientes às células do carcinoma, aumentando a tradução de HIF1A e apoiando a angiogênese.[92] O mTOR também ajuda em outra adaptação metabólica das células cancerosas para apoiar sua maior taxa de crescimento - ativação do metabolismo glicolítico. Akt2, um substrato de mTOR, especificamente de mTORC2, aumenta a expressão da enzima glicolítica PKM2, contribuindo assim para o efeito Warburg.[93]
Distúrbios do sistema nervoso central / Função cerebral
[editar | editar código-fonte]Autismo
[editar | editar código-fonte]O mTOR está implicado na falha de um mecanismo de "poda" das sinapses excitatórias nos transtornos do espectro autista.[94]
Doença de Alzheimer
[editar | editar código-fonte]A sinalização mTOR se cruza com a patologia da doença de Alzheimer (DA) em vários aspectos, sugerindo sua possível função como contribuinte para a progressão da doença. Em geral, as descobertas demonstram a hiperatividade da sinalização mTOR nos cérebros da DA. Por exemplo, estudos post-mortem de cérebros humanos com DA revelam desregulação em PTEN, Akt, S6K e mTOR.[95][96][97] A sinalização mTOR parece estar intimamente relacionada à presença de beta amiloide solúvel (Aβ) e proteínas tau, que se agregam e formam duas marcas registradas da doença, placas Aβ e emaranhados neurofibrilares, respectivamente.[98] Estudos in vitro demonstraram que o Aβ é um ativador da via PI3K/AKT, que, por sua vez, ativa o mTOR.[99] Além disso, a aplicação de Aβ em células N2K aumenta a expressão de p70S6K, um alvo a jusante de mTOR conhecido por ter maior expressão em neurônios que eventualmente desenvolvem emaranhados neurofibrilares.[100][101] Células de ovário de hamster chinês transfectadas com a mutação 7PA2 da DA familiar também apresentam maior atividade de mTOR em comparação com os controles, e a hiperatividade é bloqueada com o uso de um inibidor de gama-secretase.[102][103] Esses estudos in vitro sugerem que o aumento das concentrações de Aβ aumenta a sinalização de mTOR; entretanto, acredita-se que concentrações citotóxicas significativamente grandes de Aβ diminuam a sinalização de mTOR.[104]
Consistente com os dados observados in vitro, a atividade da mTOR e a p70S6K ativada demonstraram estar significativamente aumentadas no córtex e no hipocampo de modelos animais de DA em comparação com os controles.[103][105] A remoção farmacológica ou genética de Aβ em modelos animais de DA elimina a interrupção da atividade normal de mTOR, apontando para o envolvimento direto de Aβ na sinalização de mTOR. Além disso, ao injetar oligômeros de Aβ nos hipocampos de camundongos normais, observa-se hiperatividade de mTOR.[105] As deficiências cognitivas características da DA parecem ser mediadas pela fosforilação do PRAS-40, que se desprende e permite a hiperatividade do mTOR quando é fosforilado; a inibição da fosforilação do PRAS-40 evita a hiperatividade do mTOR induzida pelo Aβ.[105][106][107] Considerando esses achados, a via de sinalização do mTOR parece ser um mecanismo de toxicidade induzida pelo Aβ na DA.
A hiperfosforilação das proteínas tau em emaranhados neurofibrilares é uma das marcas registradas da DA. Foi demonstrado que a ativação do p70S6K promove a formação de emaranhados, bem como a hiperatividade do mTOR por meio do aumento da fosforilação e da redução da desfosforilação.[100][108][109][110] Também foi proposto que o mTOR contribui para a patologia da tau ao aumentar a tradução da tau e de outras proteínas.[111]
A plasticidade sináptica é um dos principais contribuintes para o aprendizado e a memória, dois processos que são gravemente prejudicados em pacientes com DA. O controle da tradução, ou a manutenção da homeostase proteica, demonstrou ser essencial para a plasticidade neural e é regulado pelo mTOR.[103][112][113][114][115] Tanto a superprodução quanto a subprodução de proteínas por meio da atividade do mTOR parecem contribuir para o aprendizado e a memória prejudicados. Além disso, considerando que os déficits resultantes da hiperatividade do mTOR podem ser aliviados pelo tratamento com rapamicina, é possível que o mTOR desempenhe um papel importante ao afetar o funcionamento cognitivo por meio da plasticidade sináptica.[99][116] Outras evidências da atividade do mTOR na neurodegeneração vêm de descobertas recentes que demonstram que o eIF2α-P, um alvo a montante da via do mTOR, medeia a morte celular em doenças priônicas por meio da inibição sustentada da tradução.[117]
Algumas evidências também apontam para o papel do mTOR na redução da depuração de Aβ. O mTOR é um regulador negativo da autofagia;[118] portanto, a hiperatividade na sinalização do mTOR deve reduzir a depuração de Aβ no cérebro da DA. As interrupções na autofagia podem ser uma fonte potencial de patogênese em doenças de desdobramento de proteínas, incluindo a DA.[119][120][121][122][123][124] Estudos usando modelos de camundongos da doença de Huntington demonstram que o tratamento com rapamicina facilita a depuração de agregados de huntingtina.[125][126] Talvez o mesmo tratamento possa ser útil na depuração de depósitos de Aβ também.
Doenças linfoproliferativas
[editar | editar código-fonte]As vias mTOR hiperativas foram identificadas em determinadas doenças linfoproliferativas, como a síndrome linfoproliferativa autoimune (ALPS),[127] a doença de Castleman multicêntrica,[128] e o doença linfoproliferativa pós-transplante (PTLD).[129]
Síntese de proteínas e crescimento celular
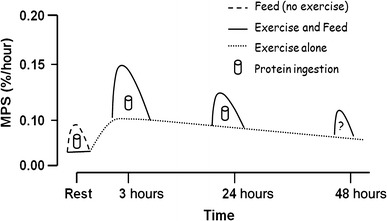
[editar | editar código-fonte]A ativação do mTORC1 é necessária para a síntese de proteínas musculares miofibrilares e hipertrofia do músculo esquelético em humanos em resposta ao exercício físico e à ingestão de determinados aminoácidos ou derivados de aminoácidos. A inativação persistente da sinalização do mTORC1 no músculo esquelético facilita a perda de massa muscular e força durante a perda de massa muscular na velhice, caquexia por câncer e atrofia muscular por inatividade física.[130][131][132] A ativação de mTORC2 parece mediar o crescimento de neuritos em células Neuro2A de camundongo diferenciadas.[133] A ativação intermitente de mTOR em neurônios pré-frontais por β-hidroxi β-metilbutirato inibe o declínio cognitivo relacionado à idade associado à poda dendrítica em animais, que é um fenômeno também observado em humanos.[134]
• PA: fosfatídeo
• mTOR: alvo mecanístico da rapamicina
• AMP: monofosfato de adenosina
• ATP: trifosfato de adenosina
• AMPK: proteína quinase ativada por AMP
• PGC‐1α: coativador-1α do receptor gama ativado por proliferador de peroxissoma
• S6K1: p70S6 quinase
• 4EBP1: proteína 1 de ligação ao fator de iniciação da tradução eucariótica 4E
• eIF4E: fator de iniciação da tradução eucariótica 4E
• RPS6: proteína ribossômica S6
• eEF2: fator de alongamento eucariótico 2
• RE: treino de força; EE: treino de resistência
• Myo: miofibrilha; Mito: mitocôndrial
• AA: aminoácido
• HMB: Ácido β-hidroxi-β-metilbutírico
• ↑ representa a ativação
• Τ representa a inibição
Danos lisossômicos inibem a mTOR e induzem a autofagia
[editar | editar código-fonte]O mTORC1 ativo está posicionado nos lisossomos. A mTOR é inibida quando a membrana lisossômica é danificada por vários agentes exógenos ou endógenos, como bactérias invasoras, substâncias químicas permeáveis à membrana que produzem produtos osmoticamente ativos (esse tipo de lesão pode ser modelado usando precursores dipeptídicos permeáveis à membrana que se polimerizam nos lisossomos), agregados de proteína amiloide (consulte a seção acima sobre a doença de Alzheimer) e inclusões orgânicas ou inorgânicas citoplasmáticas, incluindo cristais de ácido úrico e sílica cristalina. O processo de inativação do mTOR após a lise da membrana/endomembrana é mediado pelo complexo proteico denominado GALTOR. No centro do GALTOR está a galectina-8, um membro da superfamília de ligação a β-galactosídeos das lectinas citosólicas denominadas galectinas, que reconhece o dano à membrana lisossômica ligando-se aos glicanos expostos no lado luminal da endomembrana delimitadora. Após o dano à membrana, a galectina-8, que normalmente se associa ao mTOR em condições homeostáticas, não interage mais com o mTOR, mas agora se liga ao SLC38A9, RRAGA/RRAGB e LAMTOR1, inibindo a função de troca de nucleotídeos de guanina do Ragulator (complexo LAMTOR1-5).[136]
O TOR é um regulador negativo da autofagia em geral, melhor estudado durante a resposta à fome,[137][138][139][140][141] que é uma resposta metabólica. No entanto, durante o dano lisossômico, a inibição da mTOR ativa a resposta da autofagia em sua função de controle de qualidade, levando ao processo denominado lisofagia[142] que remove os lisossomos danificados. Nesse estágio, outra galectina, a galectina-3, interage com o TRIM16 para orientar a autofagia seletiva de lisossomos danificados.[143][144] O TRIM16 reúne ULK1 e os principais componentes (Beclin 1 e ATG16L1) de outros complexos (Beclin 1-VPS34-ATG14 e ATG16L1-ATG5-ATG12) iniciando a autofagia,[144] muitos deles sob controle negativo do mTOR diretamente, como o complexo ULK1-ATG13,[139][140][141] ou indiretamente, como os componentes da PI3K de classe III (Beclin 1, ATG14 e VPS34), uma vez que dependem da ativação de fosforilações por ULK1 quando não são inibidos por mTOR. Esses componentes que impulsionam a autofagia estão física e funcionalmente ligados uns aos outros, integrando todos os processos necessários para a formação de autofagossomos:
- o complexo ULK1-ATG13-FIP200/RB1CC1 associa-se à maquinaria de conjugação LC3B/GABARAP por meio de interações diretas entre FIP200/RB1CC1 e ATG16L1,[145][146][147]
- o complexo ULK1-ATG13-FIP200/RB1CC1 associa-se à Beclin 1-VPS34-ATG14 por meio de interações diretas entre o domínio HORMA de ATG13 e ATG14,[148]
- ATG16L1 interage com WIPI2, que se liga a PI3P, o produto enzimático da PI3K classe III Beclin 1-VPS34-ATG14.[149] Assim, a inativação da mTOR, iniciada por GALTOR após o dano lisossômico, mais uma ativação simultânea via galectina-9 (que também reconhece a violação da membrana lisossômica) da AMPK[136] que fosforila e ativa diretamente os principais componentes (ULK1,[150] Beclin 1[151]) dos sistemas de autofagia listados acima e inativa ainda mais o mTORC1,[152][153] permite uma forte indução de autofagia e remoção autofágica de lisossomos danificados.
Além disso, vários tipos de eventos de ubiquitinação são paralelos e complementam os processos acionados por galectina: A ubiquitinação de TRIM16-ULK1-Beclin-1 estabiliza esses complexos para promover a ativação da autofagia, conforme descrito acima.[144] O ATG16L1 tem uma afinidade de ligação intrínseca para a ubiquitina[147]); Considerando que a ubiquitinação por uma ubiquitina ligase específica de glicoproteína dotada de FBXO27 de várias proteínas de membrana lisossômica glicosiladas expostas a danos, como LAMP1, LAMP2, GNS/N-acetilglucosamina-6-sulfatase, TSPAN6/tetraspanina-6, PSAP/prosaposina e TMEM192/proteína transmembrana 192[154] podem contribuir para a execução da lisofagia por meio de receptores autofágicos, como p62/SQSTM1, que é recrutado durante a lisofagia,[147] ou outras funções a serem determinadas.
Esclerodermia
[editar | editar código-fonte]A esclerodermia, também conhecida como esclerose sistêmica, é uma doença autoimune sistêmica crônica caracterizada pelo endurecimento (esclero) da pele (derma) que afeta órgãos internos em suas formas mais graves.[155][156] O mTOR desempenha um papel nas doenças fibróticas e na autoimunidade, e o bloqueio da via mTORC está sendo investigado como um tratamento para a esclerodermia.[5]
Inibidores de mTOR como terapias
[editar | editar código-fonte]Transplante
[editar | editar código-fonte]Os inibidores de mTOR, por exemplo, a rapamicina, já são usados para evitar a rejeição de transplantes.
Doença de depósito de glicogênio
[editar | editar código-fonte]Alguns artigos relataram que a rapamicina pode inibir a mTORC1, de modo que a fosforilação da GS (glicogênio sintase) pode ser aumentada no músculo esquelético. Essa descoberta representa uma possível nova abordagem terapêutica para doenças de armazenamento de glicogênio que envolvem o acúmulo de glicogênio no músculo.
Anticâncer
[editar | editar código-fonte]Existem dois inibidores primários de mTOR usados no tratamento de cânceres humanos, o temsirolimo e o everolimo. Os inibidores de mTOR têm sido usados no tratamento de diversas malignidades, incluindo carcinoma de células renais (temsirolimo) e câncer de pâncreas, câncer de mama e carcinoma de células renais (everolimo).[157] O mecanismo completo desses agentes não está claro, mas acredita-se que eles funcionem prejudicando a angiogênese do tumor e causando prejuízo na transição G1/S.[158]
Antienvelhecimento
[editar | editar código-fonte]Os inibidores de mTOR podem ser úteis para tratar/prevenir várias condições associadas à idade,[159] incluindo doenças neurodegenerativas, como a doença de Alzheimer e a doença de Parkinson.[160] Após um tratamento de curto prazo com os inibidores de mTOR dactolisib e everolimo, em idosos (65 anos ou mais), os indivíduos tratados tiveram um número reduzido de infecções ao longo de um ano.[161]
Foi relatado que vários compostos naturais, incluindo galato de epigalocatequina (EGCG), cafeína, curcumina, berberina, quercetina, resveratrol e pterostilbeno, inibem a mTOR quando aplicados a células isoladas em cultura.[162][163][164] Até o momento, não há evidências de alta qualidade de que essas substâncias inibam a sinalização da mTOR ou prolonguem a vida útil quando tomadas como suplementos alimentares por seres humanos, apesar dos resultados encorajadores em animais como moscas-das-frutas e camundongos. Vários testes estão em andamento.[165][166][170].
Interações
[editar | editar código-fonte]Foi demonstrado que o alvo mecânico da rapamicina interage com:[167]
- ABL1[168]
- AKT1[48][169][170]
- IGF-IR[8]
- Receptor de insulina[8]
- CLIP1[171]
- EIF3F[172]
- EIF4EBP1[41][173][174][175][176][177][178][179]
- FKBP1A[180][46][181][182][183][184]
- GPHN[185]
- KIAA1303[180][41][45][46][81][173][174][175][186][187][188][189][190][191][192][193][194][195][196][197]
- PRKCD[198]
- RHEB[176][199][200][201]
- RICTOR[180][45][46][188][194][196][197]
- RPS6KB1[41][174][176][177][178][193][196][202][203][204][205][206][207][208][209]
- STAT1[210]
- STAT3[211][212]
- TPCN1, TPCN2[213]
- UBQLN1[214]
Referências
- ↑ Sabers CJ, Martin MM, Brunn GJ, Williams JM, Dumont FJ, Wiederrecht G, Abraham RT (Jan 1995). «Isolation of a Protein Target of the FKBP12-Rapamycin Complex in Mammalian Cells». J. Biol. Chem. 270 (2): 815–22. PMID 7822316. doi:10.1074/jbc.270.2.815

- ↑ a b c d e Brown EJ, Albers MW, Shin TB, Ichikawa K, Keith CT, Lane WS, Schreiber SL (Junho de 1994). «A mammalian protein targeted by G1-arresting rapamycin-receptor complex». Nature. 369 (6483): 756–8. Bibcode:1994Natur.369..756B. PMID 8008069. doi:10.1038/369756a0
- ↑ a b c d e Sabatini DM, Erdjument-Bromage H, Lui M, Tempst P, Snyder SH (Julho de 1994). «RAFT1: a mammalian protein that binds to FKBP12 in a rapamycin-dependent fashion and is homologous to yeast TORs». Cell. 78 (1): 35–43. PMID 7518356. doi:10.1016/0092-8674(94)90570-3
- ↑ a b c Sabers CJ, Martin MM, Brunn GJ, Williams JM, Dumont FJ, Wiederrecht G, Abraham RT (Janeiro de 1995). «Isolation of a protein target of the FKBP12-rapamycin complex in mammalian cells». The Journal of Biological Chemistry. 270 (2): 815–22. PMID 7822316. doi:10.1074/jbc.270.2.815

- ↑ a b Mitra A, Luna JI, Marusina AI, Merleev A, Kundu-Raychaudhuri S, Fiorentino D, Raychaudhuri SP, Maverakis E (Novembro de 2015). «Dual mTOR Inhibition Is Required to Prevent TGF-β-Mediated Fibrosis: Implications for Scleroderma». The Journal of Investigative Dermatology. 135 (11): 2873–6. PMC 4640976
 . PMID 26134944. doi:10.1038/jid.2015.252
. PMID 26134944. doi:10.1038/jid.2015.252
- ↑ a b c d e Lipton JO, Sahin M (Outubro de 2014). «The neurology of mTOR». Neuron. 84 (2): 275–291. PMC 4223653
 . PMID 25374355. doi:10.1016/j.neuron.2014.09.034.
. PMID 25374355. doi:10.1016/j.neuron.2014.09.034. A via de sinalização mTOR atua como um integrador de sistemas moleculares para apoiar as interações celulares e do organismo com o ambiente. A via da mTOR regula a homeostase influenciando diretamente a síntese de proteínas, a transcrição, a autofagia, o metabolismo e a biogênese e manutenção de organelas. Não é de surpreender, portanto, que a sinalização mTOR esteja envolvida em toda a hierarquia da função cerebral, incluindo a proliferação de células-tronco neurais, a montagem e a manutenção de circuitos, a plasticidade dependente da experiência e a regulação de comportamentos complexos como alimentação, sono e ritmos circadianos...
Figure 1: Domain structure of the mTOR kinase and components of mTORC1 and mTORC2 Figure 2: The mTOR Signaling Pathway
A função da mTOR é mediada por dois grandes complexos bioquímicos definidos por sua respectiva composição proteica e foram amplamente revisados em outros lugares (Dibble e Manning, 2013; Laplante e Sabatini, 2012 - Figura 1B). Em resumo, são comuns ao complexo mTOR 1 (mTORC1) e ao complexo mTOR 2 (mTORC2): o próprio mTOR, a proteína 8 letal de mamíferos com sec13 (mLST8; também conhecida como GβL) e a proteína de interação com mTOR contendo domínio DEP inibitório (DEPTOR). Específica para mTORC1 é a proteína associada ao regulador do alvo de rapamicina em mamíferos (Raptor) e substrato de Akt rico em prolina de 40 kDa (PRAS40) (Kim et al., 2002; Laplante e Sabatini, 2012). O Raptor é essencial para a atividade do mTORC1. O complexo mTORC2 inclui o companheiro insensível à rapamicina do mTOR (Rictor), a proteína 1 (mSIN1) que interage com a MAP quinase ativada por estresse em mamíferos e as proteínas observadas com o rictor 1 e 2 (PROTOR 1 e 2) (Jacinto et al., 2006; Jacinto et al., 2004; Pearce et al., 2007; Sarbassov et al., 2004 - Figura 1B). Rictor e mSIN1 são essenciais para a função de mTORC2. - ↑ a b c Hay N, Sonenberg N (Agosto de 2004). «Upstream and downstream of mTOR». Genes & Development. 18 (16): 1926–45. PMID 15314020. doi:10.1101/gad.1212704

- ↑ a b c d Yin Y, Hua H, Li M, Liu S, Kong Q, Shao T, Wang J, Luo Y, Wang Q, Luo T, Jiang Y (Janeiro de 2016). «mTORC2 promotes type I insulin-like growth factor receptor and insulin receptor activation through the tyrosine kinase activity of mTOR». Cell Research. 26 (1): 46–65. PMC 4816127
 . PMID 26584640. doi:10.1038/cr.2015.133
. PMID 26584640. doi:10.1038/cr.2015.133
- ↑ Jacinto E, Loewith R, Schmidt A, Lin S, Rüegg MA, Hall A, Hall MN (Novembro de 2004). «Mammalian TOR complex 2 controls the actin cytoskeleton and is rapamycin insensitive». Nature Cell Biology. 6 (11): 1122–8. PMID 15467718. doi:10.1038/ncb1183
- ↑ Sehgal, S. N.; Baker, H.; VéZina, Claude (1975). «Rapamycin (AY-22,989), a new antifungal antibiotic. II. Fermentation, isolation and characterization.». The Journal of Antibiotics (em inglês). 28 (10): 727–732. ISSN 0021-8820. doi:10.7164/antibiotics.28.727
- ↑ Powers, Ted (1 de novembro de 2022). Kellogg, Doug, ed. «The origin story of rapamycin: systemic bias in biomedical research and cold war politics». Molecular Biology of the Cell (em inglês). 33 (13). ISSN 1059-1524. PMID 36228182. doi:10.1091/mbc.E22-08-0377
- ↑ a b c Heitman J, Movva NR, Hall MN (Agosto de 1991). «Targets for cell cycle arrest by the immunosuppressant rapamycin in yeast». Science. 253 (5022): 905–9. Bibcode:1991Sci...253..905H. PMID 1715094. doi:10.1126/science.1715094
- ↑ Kunz J, Henriquez R, Schneider U, Deuter-Reinhard M, Movva NR, and Hall MN (Maio de 1993). «Target of rapamycin in yeast, TOR2, is an essential phosphatidylinositol kinase homolog required for G1 progression». Cell. 73 (3): 585–596. PMID 8387896. doi:10.1016/0092-8674(93)90144-F
- ↑ Cafferkey R, Young PR, McLaughlin MM, Bergsma DJ, Koltin Y, Sathe GM, Faucette L, Eng WK, Johnson RK, Livi GP (Outubro de 1993). «Dominant missense mutations in a novel yeast protein related to mammalian phosphatidylinositol 3-kinase and VPS34 abrogate rapamycin cytotoxicity». Mol Cell Biol. 13 (10): 6012–23. PMC 364661
 . PMID 8413204. doi:10.1128/MCB.13.10.6012
. PMID 8413204. doi:10.1128/MCB.13.10.6012
- ↑ a b Magnuson B, Ekim B, Fingar DC (Janeiro de 2012). «Regulation and function of ribosomal protein S6 kinase (S6K) within mTOR signaling networks». The Biochemical Journal. 441 (1): 1–21. PMID 22168436. doi:10.1042/BJ20110892
- ↑ a b Abraham RT, Wiederrecht GJ (1996). «Immunopharmacology of rapamycin». Annual Review of Immunology. 14: 483–510. PMID 8717522. doi:10.1146/annurev.immunol.14.1.483
- ↑ Bierer BE, Mattila PS, Standaert RF, Herzenberg LA, Burakoff SJ, Crabtree G, Schreiber SL (Dezembro de 1990). «Two distinct signal transmission pathways in T lymphocytes are inhibited by complexes formed between an immunophilin and either FK506 or rapamycin». Proceedings of the National Academy of Sciences of the United States of America. 87 (23): 9231–5. Bibcode:1990PNAS...87.9231B. PMC 55138
 . PMID 2123553. doi:10.1073/pnas.87.23.9231
. PMID 2123553. doi:10.1073/pnas.87.23.9231
- ↑ Bierer BE, Somers PK, Wandless TJ, Burakoff SJ, Schreiber SL (Outubro de 1990). «Probing immunosuppressant action with a nonnatural immunophilin ligand». Science. 250 (4980): 556–9. Bibcode:1990Sci...250..556B. PMID 1700475. doi:10.1126/science.1700475
- ↑ Dumont FJ, Melino MR, Staruch MJ, Koprak SL, Fischer PA, Sigal NH (Fevereiro de 1990). «The immunosuppressive macrolides FK-506 and rapamycin act as reciprocal antagonists in murine T cells». J Immunol. 144 (4): 1418–24. PMID 1689353. doi:10.4049/jimmunol.144.4.1418

- ↑ Dumont FJ, Staruch MJ, Koprak SL, Melino MR, Sigal NH (Janeiro de 1990). «Distinct mechanisms of suppression of murine T cell activation by the related macrolides FK-506 and rapamycin». J Immunol. 144 (1): 251–8. PMID 1688572. doi:10.4049/jimmunol.144.1.251

- ↑ Harding MW, Galat A, Uehling DE, Schreiber SL (Outubro de 1989). «A receptor for the immunosuppressant FK506 is a cis-trans peptidyl-prolyl isomerase». Nature. 341 (6244): 758–60. Bibcode:1989Natur.341..758H. PMID 2477715. doi:10.1038/341758a0
- ↑ Fretz H, Albers MW, Galat A, Standaert RF, Lane WS, Burakoff SJ, Bierer BE, Schreiber SL (Fevereiro de 1991). «Rapamycin and FK506 binding proteins (immunophilins)». Journal of the American Chemical Society. 113 (4): 1409–1411. doi:10.1021/ja00004a051
- ↑ Liu J, Farmer JD, Lane WS, Friedman J, Weissman I, Schreiber SL (Agosto de 1991). «Calcineurin is a common target of cyclophilin-cyclosporin A and FKBP-FK506 complexes». Cell. 66 (4): 807–15. PMID 1715244. doi:10.1016/0092-8674(91)90124-H
- ↑ Kunz J, Henriquez R, Schneider U, Deuter-Reinhard M, Movva NR, and Hall MN (Maio de 1993). «Target of rapamycin in yeast, TOR2, is an essential phosphatidylinositol kinase homolog required for G1 progression». Cell. 73 (3): 585–596. PMID 8387896. doi:10.1016/0092-8674(93)90144-F
- ↑ Heitman J (Novembro de 2015). «On the discovery of TOR as the target of rapamycin». PLOS Pathogens. 11 (11): e1005245. PMC 4634758
 . PMID 26540102. doi:10.1371/journal.ppat.1005245
. PMID 26540102. doi:10.1371/journal.ppat.1005245
- ↑ Kennedy BK, Lamming DW (2016). «The Mechanistic Target of Rapamycin: The Grand ConducTOR of Metabolism and Aging». Cell Metabolism. 23 (6): 990–1003. PMC 4910876
 . PMID 27304501. doi:10.1016/j.cmet.2016.05.009
. PMID 27304501. doi:10.1016/j.cmet.2016.05.009
- ↑ «Symbol report for MTOR». HGNC data for MTOR. HUGO Gene Nomenclature Committee. 1º de setembro de 2020. Consultado em 17 de dezembro de 2020
- ↑ Tokunaga C, Yoshino K, Yonezawa K (Janeiro de 2004). «mTOR integrates amino acid- and energy-sensing pathways». Biochemical and Biophysical Research Communications. 313 (2): 443–6. PMID 14684182. doi:10.1016/j.bbrc.2003.07.019
- ↑ Wipperman MF, Montrose DC, Gotto AM, Hajjar DP (2019). «Mammalian Target of Rapamycin: A Metabolic Rheostat for Regulating Adipose Tissue Function and Cardiovascular Health». The American Journal of Pathology. 189 (3): 492–501. PMC 6412382
 . PMID 30803496. doi:10.1016/j.ajpath.2018.11.013
. PMID 30803496. doi:10.1016/j.ajpath.2018.11.013
- ↑ Beevers CS, Li F, Liu L, Huang S (Agosto de 2006). «Curcumin inhibits the mammalian target of rapamycin-mediated signaling pathways in cancer cells». International Journal of Cancer. 119 (4): 757–64. PMID 16550606. doi:10.1002/ijc.21932
- ↑ Kennedy BK, Lamming DW (Junho de 2016). «The Mechanistic Target of Rapamycin: The Grand ConducTOR of Metabolism and Aging». Cell Metabolism. 23 (6): 990–1003. PMC 4910876
 . PMID 27304501. doi:10.1016/j.cmet.2016.05.009
. PMID 27304501. doi:10.1016/j.cmet.2016.05.009
- ↑ Huang S, Houghton PJ (Dezembro de 2001). «Mechanisms of resistance to rapamycins». Drug Resistance Updates. 4 (6): 378–91. PMID 12030785. doi:10.1054/drup.2002.0227
- ↑ a b Huang S, Bjornsti MA, Houghton PJ (2003). «Rapamycins: mechanism of action and cellular resistance». Cancer Biology & Therapy. 2 (3): 222–32. PMID 12878853. doi:10.4161/cbt.2.3.360

- ↑ a b Ingargiola C, Turqueto Duarte G, Robaglia C, Leprince AS, Meyer C (Outubro de 2020). «The Plant Target of Rapamycin: A Conduc TOR of Nutrition and Metabolism in Photosynthetic Organisms». Genes. 11 (11). 1285 páginas. PMC 7694126
 . PMID 33138108. doi:10.3390/genes11111285
. PMID 33138108. doi:10.3390/genes11111285
- ↑ a b Shi L, Wu Y, Sheen J (Julho de 2018). «TOR signaling in plants: conservation and innovation». Development. 145 (13). PMC 6053665
 . PMID 29986898. doi:10.1242/dev.160887
. PMID 29986898. doi:10.1242/dev.160887
- ↑ Xiong Y, Sheen J (Fevereiro de 2014). «The role of target of rapamycin signaling networks in plant growth and metabolism». Plant Physiology. 164 (2): 499–512. PMC 3912084
 . PMID 24385567. doi:10.1104/pp.113.229948
. PMID 24385567. doi:10.1104/pp.113.229948
- ↑ Wullschleger S, Loewith R, Hall MN (Fevereiro de 2006). «TOR signaling in growth and metabolism». Cell. 124 (3): 471–84. PMID 16469695. doi:10.1016/j.cell.2006.01.016

- ↑ Betz C, Hall MN (Novembro de 2013). «Where is mTOR and what is it doing there?». The Journal of Cell Biology. 203 (4): 563–74. PMC 3840941
 . PMID 24385483. doi:10.1083/jcb.201306041
. PMID 24385483. doi:10.1083/jcb.201306041
- ↑ Groenewoud MJ, Zwartkruis FJ (Agosto de 2013). «Rheb and Rags come together at the lysosome to activate mTORC1». Biochemical Society Transactions. 41 (4): 951–5. PMID 23863162. doi:10.1042/bst20130037
- ↑ Efeyan A, Zoncu R, Sabatini DM (Setembro de 2012). «Amino acids and mTORC1: from lysosomes to disease». Trends in Molecular Medicine. 18 (9): 524–33. PMC 3432651
 . PMID 22749019. doi:10.1016/j.molmed.2012.05.007
. PMID 22749019. doi:10.1016/j.molmed.2012.05.007
- ↑ a b c d e f Kim DH, Sarbassov DD, Ali SM, King JE, Latek RR, Erdjument-Bromage H, Tempst P, Sabatini DM (Julho de 2002). «mTOR interacts with raptor to form a nutrient-sensitive complex that signals to the cell growth machinery». Cell. 110 (2): 163–75. PMID 12150925. doi:10.1016/S0092-8674(02)00808-5

- ↑ Kim DH, Sarbassov DD, Ali SM, Latek RR, Guntur KV, Erdjument-Bromage H, Tempst P, Sabatini DM (Abril de 2003). «GbetaL, a positive regulator of the rapamycin-sensitive pathway required for the nutrient-sensitive interaction between raptor and mTOR». Molecular Cell. 11 (4): 895–904. PMID 12718876. doi:10.1016/S1097-2765(03)00114-X

- ↑ Fang Y, Vilella-Bach M, Bachmann R, Flanigan A, Chen J (Novembro de 2001). «Phosphatidic acid-mediated mitogenic activation of mTOR signaling». Science. 294 (5548): 1942–5. Bibcode:2001Sci...294.1942F. PMID 11729323. doi:10.1126/science.1066015
- ↑ Bond P (Março de 2016). «Regulation of mTORC1 by growth factors, energy status, amino acids and mechanical stimuli at a glance». J. Int. Soc. Sports Nutr. 13. 8 páginas. PMC 4774173
 . PMID 26937223. doi:10.1186/s12970-016-0118-y
. PMID 26937223. doi:10.1186/s12970-016-0118-y
- ↑ a b c Frias MA, Thoreen CC, Jaffe JD, Schroder W, Sculley T, Carr SA, Sabatini DM (Setembro de 2006). «mSin1 is necessary for Akt/PKB phosphorylation, and its isoforms define three distinct mTORC2s». Current Biology. 16 (18): 1865–70. Bibcode:2006CBio...16.1865F. PMID 16919458. doi:10.1016/j.cub.2006.08.001

- ↑ a b c d e Sarbassov DD, Ali SM, Kim DH, Guertin DA, Latek RR, Erdjument-Bromage H, Tempst P, Sabatini DM (Setembro de 2006). «Rictor, a novel binding partner of mTOR, defines a rapamycin-insensitive and raptor-independent pathway that regulates the cytoskeleton». Current Biology. 14 (14): 1296–302. Bibcode:2004CBio...14.1296D. PMID 15268862. doi:10.1016/j.cub.2004.06.054

- ↑ Betz C, Stracka D, Prescianotto-Baschong C, Frieden M, Demaurex N, Hall MN (Julho de 2013). «Feature Article: mTOR complex 2-Akt signaling at mitochondria-associated endoplasmic reticulum membranes (MAM) regulates mitochondrial physiology». Proceedings of the National Academy of Sciences of the United States of America. 110 (31): 12526–34. PMC 3732980
 . PMID 23852728. doi:10.1073/pnas.1302455110
. PMID 23852728. doi:10.1073/pnas.1302455110
- ↑ a b Sarbassov DD, Guertin DA, Ali SM, Sabatini DM (Fevereiro de 2005). «Phosphorylation and regulation of Akt/PKB by the rictor-mTOR complex». Science. 307 (5712): 1098–101. Bibcode:2005Sci...307.1098S. PMID 15718470. doi:10.1126/science.1106148
- ↑ Stephens L, Anderson K, Stokoe D, Erdjument-Bromage H, Painter GF, Holmes AB, Gaffney PR, Reese CB, McCormick F, Tempst P, Coadwell J, Hawkins PT (Fevereiro de 2005). «Protein kinase B kinases that mediate phosphatidylinositol 3,4,5-trisphosphate-dependent activation of protein kinase B». Science. 279 (5351): 710–4. Bibcode:1998Sci...279..710S. PMID 9445477. doi:10.1126/science.279.5351.710
- ↑ Carosi JM, Fourrier C, Bensalem J, Sargeant TJ (2022). «The mTOR-lysosome axis at the centre of ageing». FEBS Open Bio. 12 (4): 739–757. PMC 8972043
 . PMID 34878722. doi:10.1002/2211-5463.13347
. PMID 34878722. doi:10.1002/2211-5463.13347
- ↑ Zhou S, Tang X, Chen H (2018). «Sirtuins and Insulin Resistance». Frontiers in Endocrinology. 9. 748 páginas. PMC 6291425
 . PMID 30574122. doi:10.3389/fendo.2018.00748
. PMID 30574122. doi:10.3389/fendo.2018.00748
- ↑ Baechle JJ, Chen N, Winer DA (2023). «Chronic inflammation and the hallmarks of aging». Molecular Metabolism. 74. 101755 páginas. doi:10.1016/j.molmet.2023.101755
- ↑ a b Lamming DW, Ye L, Katajisto P, Goncalves MD, Saitoh M, Stevens DM, Davis JG, Salmon AB, Richardson A, Ahima RS, Guertin DA, Sabatini DM, Baur JA (Março de 2012). «Rapamycin-induced insulin resistance is mediated by mTORC2 loss and uncoupled from longevity». Science. 335 (6076): 1638–43. Bibcode:2012Sci...335.1638L. PMC 3324089
 . PMID 22461615. doi:10.1126/science.1215135
. PMID 22461615. doi:10.1126/science.1215135
- ↑ Zinzalla V, Stracka D, Oppliger W, Hall MN (Março de 2011). «Activation of mTORC2 by association with the ribosome». Cell. 144 (5): 757–68. PMID 21376236. doi:10.1016/j.cell.2011.02.014

- ↑ Zhang F, Zhang X, Li M, Chen P, Zhang B, Guo H, Cao W, Wei X, Cao X, Hao X, Zhang N (Novembro de 2010). «mTOR complex component Rictor interacts with PKCzeta and regulates cancer cell metastasis». Cancer Research. 70 (22): 9360–70. PMID 20978191. doi:10.1158/0008-5472.CAN-10-0207

- ↑ Guertin DA, Stevens DM, Thoreen CC, Burds AA, Kalaany NY, Moffat J, Brown M, Fitzgerald KJ, Sabatini DM (Março de 2011). «Ablation in mice of the mTORC components raptor, rictor, or mLST8 reveals that mTORC2 is required for signaling to Akt-FOXO and PKCalpha, but not S6K1». Developmental Cell. 11 (6): 859–71. PMID 17141160. doi:10.1016/j.devcel.2006.10.007

- ↑ Gu Y, Lindner J, Kumar A, Yuan W, Magnuson MA (Março de 2011). «Rictor/mTORC2 is essential for maintaining a balance between beta-cell proliferation and cell size». Diabetes. 60 (3): 827–37. PMC 3046843
 . PMID 21266327. doi:10.2337/db10-1194
. PMID 21266327. doi:10.2337/db10-1194
- ↑ Lamming DW, Demirkan G, Boylan JM, Mihaylova MM, Peng T, Ferreira J, Neretti N, Salomon A, Sabatini DM, Gruppuso PA (Janeiro de 2014). «Hepatic signaling by the mechanistic target of rapamycin complex 2 (mTORC2)». FASEB Journal. 28 (1): 300–15. PMC 3868844
 . PMID 24072782. doi:10.1096/fj.13-237743
. PMID 24072782. doi:10.1096/fj.13-237743
- ↑ Kumar A, Lawrence JC, Jung DY, Ko HJ, Keller SR, Kim JK, Magnuson MA, Harris TE (Junho de 2010). «Fat cell-specific ablation of rictor in mice impairs insulin-regulated fat cell and whole-body glucose and lipid metabolism». Diabetes. 59 (6): 1397–406. PMC 2874700
 . PMID 20332342. doi:10.2337/db09-1061
. PMID 20332342. doi:10.2337/db09-1061
- ↑ Lamming DW, Mihaylova MM, Katajisto P, Baar EL, Yilmaz OH, Hutchins A, Gultekin Y, Gaither R, Sabatini DM (Outubro de 2014). «Depletion of Rictor, an essential protein component of mTORC2, decreases male lifespan». Aging Cell. 13 (5): 911–7. PMC 4172536
 . PMID 25059582. doi:10.1111/acel.12256
. PMID 25059582. doi:10.1111/acel.12256
- ↑ Feldman ME, Apsel B, Uotila A, Loewith R, Knight ZA, Ruggero D, Shokat KM (Fevereiro de 2009). «Active-site inhibitors of mTOR target rapamycin-resistant outputs of mTORC1 and mTORC2». PLOS Biology. 7 (2): e38. PMC 2637922
 . PMID 19209957. doi:10.1371/journal.pbio.1000038
. PMID 19209957. doi:10.1371/journal.pbio.1000038
- ↑ Wu JJ, Liu J, Chen EB, Wang JJ, Cao L, Narayan N, Fergusson MM, Rovira II, Allen M, Springer DA, Lago CU, Zhang S, DuBois W, Ward T, deCabo R, Gavrilova O, Mock B, Finkel T (Setembro de 2013). «Increased mammalian lifespan and a segmental and tissue-specific slowing of aging after genetic reduction of mTOR expression». Cell Reports. 4 (5): 913–20. PMC 3784301
 . PMID 23994476. doi:10.1016/j.celrep.2013.07.030
. PMID 23994476. doi:10.1016/j.celrep.2013.07.030
- ↑ Lawlor MA, Mora A, Ashby PR, Williams MR, Murray-Tait V, Malone L, Prescott AR, Lucocq JM, Alessi DR (Julho de 2002). «Essential role of PDK1 in regulating cell size and development in mice». The EMBO Journal. 21 (14): 3728–38. PMC 126129
 . PMID 12110585. doi:10.1093/emboj/cdf387
. PMID 12110585. doi:10.1093/emboj/cdf387
- ↑ Yang ZZ, Tschopp O, Baudry A, Dümmler B, Hynx D, Hemmings BA (Abril de 2004). «Physiological functions of protein kinase B/Akt». Biochemical Society Transactions. 32 (Pt 2): 350–4. PMID 15046607. doi:10.1042/BST0320350
- ↑ Nojima A, Yamashita M, Yoshida Y, Shimizu I, Ichimiya H, Kamimura N, Kobayashi Y, Ohta S, Ishii N, Minamino T (1 de janeiro de 2013). «Haploinsufficiency of akt1 prolongs the lifespan of mice». PLOS ONE. 8 (7): e69178. Bibcode:2013PLoSO...869178N. PMC 3728301
 . PMID 23935948. doi:10.1371/journal.pone.0069178
. PMID 23935948. doi:10.1371/journal.pone.0069178
- ↑ Crespo JL, Hall MN (Dezembro de 2002). «Elucidating TOR signaling and rapamycin action: lessons from Saccharomyces cerevisiae». Microbiology and Molecular Biology Reviews. 66 (4): 579–91, table of contents. PMC 134654
 . PMID 12456783. doi:10.1128/mmbr.66.4.579-591.2002
. PMID 12456783. doi:10.1128/mmbr.66.4.579-591.2002
- ↑ Peter GJ, Düring L, Ahmed A (Março de 2006). «Carbon catabolite repression regulates amino acid permeases in Saccharomyces cerevisiae via the TOR signaling pathway». The Journal of Biological Chemistry. 281 (9): 5546–52. PMID 16407266. doi:10.1074/jbc.M513842200

- ↑ a b Powers RW, Kaeberlein M, Caldwell SD, Kennedy BK, Fields S (Janeiro de 2006). «Extension of chronological life span in yeast by decreased TOR pathway signaling». Genes & Development. 20 (2): 174–84. PMC 1356109
 . PMID 16418483. doi:10.1101/gad.1381406
. PMID 16418483. doi:10.1101/gad.1381406
- ↑ a b Kaeberlein M, Powers RW, Steffen KK, Westman EA, Hu D, Dang N, Kerr EO, Kirkland KT, Fields S, Kennedy BK (Novembro de 2005). «Regulation of yeast replicative life span by TOR and Sch9 in response to nutrients». Science. 310 (5751): 1193–6. Bibcode:2005Sci...310.1193K. PMID 16293764. doi:10.1126/science.1115535
- ↑ Jia K, Chen D, Riddle DL (Agosto de 2004). «The TOR pathway interacts with the insulin signaling pathway to regulate C. elegans larval development, metabolism and life span». Development. 131 (16): 3897–906. PMID 15253933. doi:10.1242/dev.01255
- ↑ Kapahi P, Zid BM, Harper T, Koslover D, Sapin V, Benzer S (Maio de 2004). «Regulation of lifespan in Drosophila by modulation of genes in the TOR signaling pathway». Current Biology. 14 (10): 885–90. Bibcode:2004CBio...14..885K. PMC 2754830
 . PMID 15186745. doi:10.1016/j.cub.2004.03.059
. PMID 15186745. doi:10.1016/j.cub.2004.03.059
- ↑ Harrison DE, Strong R, Sharp ZD, Nelson JF, Astle CM, Flurkey K, Nadon NL, Wilkinson JE, Frenkel K, Carter CS, Pahor M, Javors MA, Fernandez E, Miller RA (Julho de 2009). «Rapamycin fed late in life extends lifespan in genetically heterogeneous mice». Nature. 460 (7253): 392–5. Bibcode:2009Natur.460..392H. PMC 2786175
 . PMID 19587680. doi:10.1038/nature08221
. PMID 19587680. doi:10.1038/nature08221
- ↑ Miller RA, Harrison DE, Astle CM, Fernandez E, Flurkey K, Han M, Javors MA, Li X, Nadon NL, Nelson JF, Pletcher S, Salmon AB, Sharp ZD, Van Roekel S, Winkleman L, Strong R (Junho de 2014). «Rapamycin-mediated lifespan increase in mice is dose and sex dependent and metabolically distinct from dietary restriction». Aging Cell. 13 (3): 468–77. PMC 4032600
 . PMID 24341993. doi:10.1111/acel.12194
. PMID 24341993. doi:10.1111/acel.12194
- ↑ Fok WC, Chen Y, Bokov A, Zhang Y, Salmon AB, Diaz V, Javors M, Wood WH, Zhang Y, Becker KG, Pérez VI, Richardson A (1 de janeiro de 2014). «Mice fed rapamycin have an increase in lifespan associated with major changes in the liver transcriptome». PLOS ONE. 9 (1): e83988. Bibcode:2014PLoSO...983988F. PMC 3883653
 . PMID 24409289. doi:10.1371/journal.pone.0083988
. PMID 24409289. doi:10.1371/journal.pone.0083988
- ↑ Arriola Apelo SI, Pumper CP, Baar EL, Cummings NE, Lamming DW (Julho de 2016). «Intermittent Administration of Rapamycin Extends the Life Span of Female C57BL/6J Mice». The Journals of Gerontology. Series A, Biological Sciences and Medical Sciences. 71 (7): 876–81. PMC 4906329
 . PMID 27091134. doi:10.1093/gerona/glw064
. PMID 27091134. doi:10.1093/gerona/glw064
- ↑ Popovich IG, Anisimov VN, Zabezhinski MA, Semenchenko AV, Tyndyk ML, Yurova MN, Blagosklonny MV (Maio de 2014). «Lifespan extension and cancer prevention in HER-2/neu transgenic mice treated with low intermittent doses of rapamycin». Cancer Biology & Therapy. 15 (5): 586–92. PMC 4026081
 . PMID 24556924. doi:10.4161/cbt.28164
. PMID 24556924. doi:10.4161/cbt.28164
- ↑ Baar EL, Carbajal KA, Ong IM, Lamming DW (Fevereiro de 2016). «Sex- and tissue-specific changes in mTOR signaling with age in C57BL/6J mice». Aging Cell. 15 (1): 155–66. PMC 4717274
 . PMID 26695882. doi:10.1111/acel.12425
. PMID 26695882. doi:10.1111/acel.12425
- ↑ Caron A, Richard D, Laplante M (Jul 2015). «The Roles of mTOR Complexes in Lipid Metabolism». Annual Review of Nutrition. 35: 321–48. PMID 26185979. doi:10.1146/annurev-nutr-071714-034355
- ↑ Cota D, Proulx K, Smith KA, Kozma SC, Thomas G, Woods SC, Seeley RJ (Maio de 2006). «Hypothalamic mTOR signaling regulates food intake». Science. 312 (5775): 927–30. Bibcode:2006Sci...312..927C. PMID 16690869. doi:10.1126/science.1124147
- ↑ a b Kriete A, Bosl WJ, Booker G (Junho de 2010). «Rule-based cell systems model of aging using feedback loop motifs mediated by stress responses». PLOS Computational Biology. 6 (6): e1000820. Bibcode:2010PLSCB...6E0820K. PMC 2887462
 . PMID 20585546. doi:10.1371/journal.pcbi.1000820
. PMID 20585546. doi:10.1371/journal.pcbi.1000820
- ↑ a b Schieke SM, Phillips D, McCoy JP, Aponte AM, Shen RF, Balaban RS, Finkel T (Setembro de 2006). «The mammalian target of rapamycin (mTOR) pathway regulates mitochondrial oxygen consumption and oxidative capacity». The Journal of Biological Chemistry. 281 (37): 27643–52. PMID 16847060. doi:10.1074/jbc.M603536200

- ↑ Yessenkyzy A, Saliev T, Zhanaliyeva M, Nurgozhin T (2020). «Polyphenols as Caloric-Restriction Mimetics and Autophagy Inducers in Aging Research». Nutrients. 12 (5). 1344 páginas. PMC 7285205
 . PMID 32397145. doi:10.3390/nu12051344
. PMID 32397145. doi:10.3390/nu12051344
- ↑ a b Laberge R, Sun Y, Orjalo AV, Patil CK, Campisi J (2015). «MTOR regulates the pro-tumorigenic senescence-associated secretory phenotype by promoting IL1A translation». Nature Cell Biology. 17 (8): 1049–1061. PMC 4691706
 . PMID 26147250. doi:10.1038/ncb3195
. PMID 26147250. doi:10.1038/ncb3195
- ↑ Wang R, Yu Z, Sunchu B, Perez VI (2017). «Rapamycin inhibits the secretory phenotype of senescent cells by a Nrf2-independent mechanism». Aging Cell. 16 (3): 564–574. PMC 5418203
 . PMID 28371119. doi:10.1111/acel.12587
. PMID 28371119. doi:10.1111/acel.12587
- ↑ Wang R, Sunchu B, Perez VI (2017). «Rapamycin and the inhibition of the secretory phenotype». Experimental Gerontology. 94: 89–92. PMID 28167236. doi:10.1016/j.exger.2017.01.026
- ↑ Weichhart T (2018). «mTOR as Regulator of Lifespan, Aging, and Cellular Senescence: A Mini-Review». Gerontology. 84 (2): 127–134. PMC 6089343
 . PMID 29190625. doi:10.1159/000484629
. PMID 29190625. doi:10.1159/000484629
- ↑ Xu K, Liu P, Wei W (Dezembro de 2014). «mTOR signaling in tumorigenesis». Biochimica et Biophysica Acta (BBA) - Reviews on Cancer. 1846 (2): 638–54. PMC 4261029
 . PMID 25450580. doi:10.1016/j.bbcan.2014.10.007
. PMID 25450580. doi:10.1016/j.bbcan.2014.10.007
- ↑ Guertin DA, Sabatini DM (Agosto de 2005). «An expanding role for mTOR in cancer». Trends in Molecular Medicine. 11 (8): 353–61. PMID 16002336. doi:10.1016/j.molmed.2005.06.007
- ↑ Pópulo H, Lopes JM, Soares P (2012). «The mTOR signalling pathway in human cancer». International Journal of Molecular Sciences. 13 (2): 1886–918. PMC 3291999
 . PMID 22408430. doi:10.3390/ijms13021886
. PMID 22408430. doi:10.3390/ijms13021886
- ↑ Easton JB, Houghton PJ (Outubro de 2006). «mTOR and cancer therapy». Oncogene. 25 (48): 6436–46. PMID 17041628. doi:10.1038/sj.onc.1209886
- ↑ Zoncu R, Efeyan A, Sabatini DM (Janeiro de 2011). «mTOR: from growth signal integration to cancer, diabetes and ageing». Nature Reviews Molecular Cell Biology. 12 (1): 21–35. PMC 3390257
 . PMID 21157483. doi:10.1038/nrm3025
. PMID 21157483. doi:10.1038/nrm3025
- ↑ Thomas GV, Tran C, Mellinghoff IK, Welsbie DS, Chan E, Fueger B, Czernin J, Sawyers CL (Janeiro de 2006). «Hypoxia-inducible factor determines sensitivity to inhibitors of mTOR in kidney cancer». Nature Medicine. 12 (1): 122–7. PMID 16341243. doi:10.1038/nm1337
- ↑ Nemazanyy I, Espeillac C, Pende M, Panasyuk G (Agosto de 2013). «Role of PI3K, mTOR and Akt2 signalling in hepatic tumorigenesis via the control of PKM2 expression». Biochemical Society Transactions. 41 (4): 917–22. PMID 23863156. doi:10.1042/BST20130034
- ↑ Tang G, Gudsnuk K, Kuo SH, Cotrina ML, Rosoklija G, Sosunov A, Sonders MS, Kanter E, Castagna C, Yamamoto A, Yue Z, Arancio O, Peterson BS, Champagne F, Dwork AJ, Goldman J, Sulzer D (Setembro de 2014). «Loss of mTOR-dependent macroautophagy causes autistic-like synaptic pruning deficits». Neuron. 83 (5): 1131–43. PMC 4159743
 . PMID 25155956. doi:10.1016/j.neuron.2014.07.040
. PMID 25155956. doi:10.1016/j.neuron.2014.07.040
- ↑ Rosner M, Hanneder M, Siegel N, Valli A, Fuchs C, Hengstschläger M (Junho de 2008). «The mTOR pathway and its role in human genetic diseases». Mutation Research. 659 (3): 284–92. PMID 18598780. doi:10.1016/j.mrrev.2008.06.001
- ↑ Li X, Alafuzoff I, Soininen H, Winblad B, Pei JJ (Agosto de 2005). «Levels of mTOR and its downstream targets 4E-BP1, eEF2, and eEF2 kinase in relationships with tau in Alzheimer's disease brain». The FEBS Journal. 272 (16): 4211–20. PMID 16098202. doi:10.1111/j.1742-4658.2005.04833.x
- ↑ Chano T, Okabe H, Hulette CM (Setembro de 2007). «RB1CC1 insufficiency causes neuronal atrophy through mTOR signaling alteration and involved in the pathology of Alzheimer's diseases». Brain Research. 1168 (1168): 97–105. PMID 17706618. doi:10.1016/j.brainres.2007.06.075
- ↑ Selkoe DJ (Setembro de 2008). «Soluble oligomers of the amyloid beta-protein impair synaptic plasticity and behavior». Behavioural Brain Research. 192 (1): 106–13. PMC 2601528
 . PMID 18359102. doi:10.1016/j.bbr.2008.02.016
. PMID 18359102. doi:10.1016/j.bbr.2008.02.016
- ↑ a b Oddo S (Janeiro de 2012). «The role of mTOR signaling in Alzheimer disease». Frontiers in Bioscience. 4 (1): 941–52. PMC 4111148
 . PMID 22202101. doi:10.2741/s310
. PMID 22202101. doi:10.2741/s310
- ↑ a b An WL, Cowburn RF, Li L, Braak H, Alafuzoff I, Iqbal K, Iqbal IG, Winblad B, Pei JJ (Agosto de 2003). «Up-regulation of phosphorylated/activated p70 S6 kinase and its relationship to neurofibrillary pathology in Alzheimer's disease». The American Journal of Pathology. 163 (2): 591–607. PMC 1868198
 . PMID 12875979. doi:10.1016/S0002-9440(10)63687-5
. PMID 12875979. doi:10.1016/S0002-9440(10)63687-5
- ↑ Zhang F, Beharry ZM, Harris TE, Lilly MB, Smith CD, Mahajan S, Kraft AS (Maio de 2009). «PIM1 protein kinase regulates PRAS40 phosphorylation and mTOR activity in FDCP1 cells». Cancer Biology & Therapy. 8 (9): 846–53. PMID 19276681. doi:10.4161/cbt.8.9.8210
- ↑ Koo EH, Squazzo SL (Julho de 1994). «Evidence that production and release of amyloid beta-protein involves the endocytic pathway». The Journal of Biological Chemistry. 269 (26): 17386–9. PMID 8021238. doi:10.1016/S0021-9258(17)32449-3

- ↑ a b c Caccamo A, Majumder S, Richardson A, Strong R, Oddo S (Abril de 2010). «Molecular interplay between mammalian target of rapamycin (mTOR), amyloid-beta, and Tau: effects on cognitive impairments». The Journal of Biological Chemistry. 285 (17): 13107–20. PMC 2857107
 . PMID 20178983. doi:10.1074/jbc.M110.100420
. PMID 20178983. doi:10.1074/jbc.M110.100420
- ↑ Lafay-Chebassier C, Paccalin M, Page G, Barc-Pain S, Perault-Pochat MC, Gil R, Pradier L, Hugon J (Julho de 2005). «mTOR/p70S6k signalling alteration by Abeta exposure as well as in APP-PS1 transgenic models and in patients with Alzheimer's disease». Journal of Neurochemistry. 94 (1): 215–25. PMID 15953364. doi:10.1111/j.1471-4159.2005.03187.x

- ↑ a b c Caccamo A, Maldonado MA, Majumder S, Medina DX, Holbein W, Magrí A, Oddo S (Março de 2011). «Naturally secreted amyloid-beta increases mammalian target of rapamycin (mTOR) activity via a PRAS40-mediated mechanism». The Journal of Biological Chemistry. 286 (11): 8924–32. PMC 3058958
 . PMID 21266573. doi:10.1074/jbc.M110.180638
. PMID 21266573. doi:10.1074/jbc.M110.180638
- ↑ Sancak Y, Thoreen CC, Peterson TR, Lindquist RA, Kang SA, Spooner E, Carr SA, Sabatini DM (Março de 2007). «PRAS40 is an insulin-regulated inhibitor of the mTORC1 protein kinase». Molecular Cell. 25 (6): 903–15. PMID 17386266. doi:10.1016/j.molcel.2007.03.003

- ↑ Wang L, Harris TE, Roth RA, Lawrence JC (Julho de 2007). «PRAS40 regulates mTORC1 kinase activity by functioning as a direct inhibitor of substrate binding». The Journal of Biological Chemistry. 282 (27): 20036–44. PMID 17510057. doi:10.1074/jbc.M702376200

- ↑ Pei JJ, Hugon J (Dezembro de 2008). «mTOR-dependent signalling in Alzheimer's disease». Journal of Cellular and Molecular Medicine. 12 (6B): 2525–32. PMC 3828871
 . PMID 19210753. doi:10.1111/j.1582-4934.2008.00509.x
. PMID 19210753. doi:10.1111/j.1582-4934.2008.00509.x
- ↑ Meske V, Albert F, Ohm TG (Janeiro de 2008). «Coupling of mammalian target of rapamycin with phosphoinositide 3-kinase signaling pathway regulates protein phosphatase 2A- and glycogen synthase kinase-3 -dependent phosphorylation of Tau». The Journal of Biological Chemistry. 283 (1): 100–9. PMID 17971449. doi:10.1074/jbc.M704292200

- ↑ Janssens V, Goris J (Fevereiro de 2001). «Protein phosphatase 2A: a highly regulated family of serine/threonine phosphatases implicated in cell growth and signalling». The Biochemical Journal. 353 (Pt 3): 417–39. PMC 1221586
 . PMID 11171037. doi:10.1042/0264-6021:3530417
. PMID 11171037. doi:10.1042/0264-6021:3530417
- ↑ Morita T, Sobue K (Outubro de 2009). «Specification of neuronal polarity regulated by local translation of CRMP2 and Tau via the mTOR-p70S6K pathway». The Journal of Biological Chemistry. 284 (40): 27734–45. PMC 2785701
 . PMID 19648118. doi:10.1074/jbc.M109.008177
. PMID 19648118. doi:10.1074/jbc.M109.008177
- ↑ Puighermanal E, Marsicano G, Busquets-Garcia A, Lutz B, Maldonado R, Ozaita A (Setembro de 2009). «Cannabinoid modulation of hippocampal long-term memory is mediated by mTOR signaling». Nature Neuroscience. 12 (9): 1152–8. PMID 19648913. doi:10.1038/nn.2369
- ↑ Tischmeyer W, Schicknick H, Kraus M, Seidenbecher CI, Staak S, Scheich H, Gundelfinger ED (Agosto de 2003). «Rapamycin-sensitive signalling in long-term consolidation of auditory cortex-dependent memory». The European Journal of Neuroscience. 18 (4): 942–50. PMID 12925020. doi:10.1046/j.1460-9568.2003.02820.x
- ↑ Hoeffer CA, Klann E (Fevereiro de 2010). «mTOR signaling: at the crossroads of plasticity, memory and disease». Trends in Neurosciences. 33 (2): 67–75. PMC 2821969
 . PMID 19963289. doi:10.1016/j.tins.2009.11.003
. PMID 19963289. doi:10.1016/j.tins.2009.11.003
- ↑ Kelleher RJ, Govindarajan A, Jung HY, Kang H, Tonegawa S (Fevereiro de 2004). «Translational control by MAPK signaling in long-term synaptic plasticity and memory». Cell. 116 (3): 467–79. PMID 15016380. doi:10.1016/S0092-8674(04)00115-1

- ↑ Ehninger D, Han S, Shilyansky C, Zhou Y, Li W, Kwiatkowski DJ, Ramesh V, Silva AJ (Agosto de 2008). «Reversal of learning deficits in a Tsc2+/- mouse model of tuberous sclerosis». Nature Medicine. 14 (8): 843–8. PMC 2664098
 . PMID 18568033. doi:10.1038/nm1788
. PMID 18568033. doi:10.1038/nm1788
- ↑ Moreno JA, Radford H, Peretti D, Steinert JR, Verity N, Martin MG, Halliday M, Morgan J, Dinsdale D, Ortori CA, Barrett DA, Tsaytler P, Bertolotti A, Willis AE, Bushell M, Mallucci GR (Maio de 2012). «Sustained translational repression by eIF2α-P mediates prion neurodegeneration». Nature. 485 (7399): 507–11. Bibcode:2012Natur.485..507M. PMC 3378208
 . PMID 22622579. doi:10.1038/nature11058
. PMID 22622579. doi:10.1038/nature11058
- ↑ Díaz-Troya S, Pérez-Pérez ME, Florencio FJ, Crespo JL (Outubro de 2008). «The role of TOR in autophagy regulation from yeast to plants and mammals». Autophagy. 4 (7): 851–65. PMID 18670193. doi:10.4161/auto.6555

- ↑ McCray BA, Taylor JP (Dezembro de 2008). «The role of autophagy in age-related neurodegeneration». Neuro-Signals. 16 (1): 75–84. PMID 18097162. doi:10.1159/000109761
- ↑ Nedelsky NB, Todd PK, Taylor JP (Dezembro de 2008). «Autophagy and the ubiquitin-proteasome system: collaborators in neuroprotection». Biochimica et Biophysica Acta (BBA) - Molecular Basis of Disease. 1782 (12): 691–9. PMC 2621359
 . PMID 18930136. doi:10.1016/j.bbadis.2008.10.002
. PMID 18930136. doi:10.1016/j.bbadis.2008.10.002
- ↑ Rubinsztein DC (Outubro de 2006). «The roles of intracellular protein-degradation pathways in neurodegeneration». Nature. 443 (7113): 780–6. Bibcode:2006Natur.443..780R. PMID 17051204. doi:10.1038/nature05291
- ↑ Oddo S (Abril de 2008). «The ubiquitin-proteasome system in Alzheimer's disease». Journal of Cellular and Molecular Medicine. 12 (2): 363–73. PMC 3822529
 . PMID 18266959. doi:10.1111/j.1582-4934.2008.00276.x
. PMID 18266959. doi:10.1111/j.1582-4934.2008.00276.x
- ↑ Li X, Li H, Li XJ (Novembro de 2008). «Intracellular degradation of misfolded proteins in polyglutamine neurodegenerative diseases». Brain Research Reviews. 59 (1): 245–52. PMC 2577582
 . PMID 18773920. doi:10.1016/j.brainresrev.2008.08.003
. PMID 18773920. doi:10.1016/j.brainresrev.2008.08.003
- ↑ Caccamo A, Majumder S, Deng JJ, Bai Y, Thornton FB, Oddo S (Outubro de 2009). «Rapamycin rescues TDP-43 mislocalization and the associated low molecular mass neurofilament instability». The Journal of Biological Chemistry. 284 (40): 27416–24. PMC 2785671
 . PMID 19651785. doi:10.1074/jbc.M109.031278
. PMID 19651785. doi:10.1074/jbc.M109.031278
- ↑ Ravikumar B, Vacher C, Berger Z, Davies JE, Luo S, Oroz LG, Scaravilli F, Easton DF, Duden R, O'Kane CJ, Rubinsztein DC (Junho de 2004). «Inhibition of mTOR induces autophagy and reduces toxicity of polyglutamine expansions in fly and mouse models of Huntington disease». Nature Genetics. 36 (6): 585–95. PMID 15146184. doi:10.1038/ng1362

- ↑ Rami A (Outubro de 2009). «Review: autophagy in neurodegeneration: firefighter and/or incendiarist?». Neuropathology and Applied Neurobiology. 35 (5): 449–61. PMID 19555462. doi:10.1111/j.1365-2990.2009.01034.x

- ↑ Völkl, Simon, et al. "Hyperactive mTOR pathway promotes lymphoproliferation and abnormal differentiation in autoimmune lymphoproliferative syndrome." Blood, The Journal of the American Society of Hematology 128.2 (2016): 227-238. https://doi.org/10.1182/blood-2015-11-685024
- ↑ Arenas, Daniel J., et al. "Increased mTOR activation in idiopathic multicentric Castleman disease." Blood 135.19 (2020): 1673-1684. https://doi.org/10.1182/blood.2019002792
- ↑ El-Salem, Mouna, et al. "Constitutive activation of mTOR signaling pathway in post-transplant lymphoproliferative disorders." Laboratory Investigation 87.1 (2007): 29-39. https://doi.org/10.1038/labinvest.3700494
- ↑ a b c Brook MS, Wilkinson DJ, Phillips BE, Perez-Schindler J, Philp A, Smith K, Atherton PJ (Janeiro 2016). «Skeletal muscle homeostasis and plasticity in youth and ageing: impact of nutrition and exercise». Acta Physiologica. 216 (1): 15–41. PMC 4843955
 . PMID 26010896. doi:10.1111/apha.12532
. PMID 26010896. doi:10.1111/apha.12532
- ↑ Brioche T, Pagano AF, Py G, Chopard A (Abril de 2016). «Muscle wasting and aging: Experimental models, fatty infiltrations, and prevention» (PDF). Molecular Aspects of Medicine. 50: 56–87. PMID 27106402. doi:10.1016/j.mam.2016.04.006
- ↑ Drummond MJ, Dreyer HC, Fry CS, Glynn EL, Rasmussen BB (Abril de 2009). «Nutritional and contractile regulation of human skeletal muscle protein synthesis and mTORC1 signaling». Journal of Applied Physiology. 106 (4): 1374–84. PMC 2698645
 . PMID 19150856. doi:10.1152/japplphysiol.91397.2008
. PMID 19150856. doi:10.1152/japplphysiol.91397.2008
- ↑ Salto R, Vílchez JD, Girón MD, Cabrera E, Campos N, Manzano M, Rueda R, López-Pedrosa JM (2015). «β-Hydroxy-β-Methylbutyrate (HMB) Promotes Neurite Outgrowth in Neuro2a Cells». PLOS ONE. 10 (8): e0135614. Bibcode:2015PLoSO..1035614S. PMC 4534402
 . PMID 26267903. doi:10.1371/journal.pone.0135614
. PMID 26267903. doi:10.1371/journal.pone.0135614
- ↑ Kougias DG, Nolan SO, Koss WA, Kim T, Hankosky ER, Gulley JM, Juraska JM (Abril de 2016). «Beta-hydroxy-beta-methylbutyrate ameliorates aging effects in the dendritic tree of pyramidal neurons in the medial prefrontal cortex of both male and female rats». Neurobiology of Aging. 40: 78–85. PMID 26973106. doi:10.1016/j.neurobiolaging.2016.01.004
- ↑ a b Phillips SM (Maio de 2014). «A brief review of critical processes in exercise-induced muscular hypertrophy». Sports Med. 44 (Suppl 1): S71–S77. PMC 4008813
 . PMID 24791918. doi:10.1007/s40279-014-0152-3
. PMID 24791918. doi:10.1007/s40279-014-0152-3
- ↑ a b Jia J, Abudu YP, Claude-Taupin A, Gu Y, Kumar S, Choi SW, Peters R, Mudd MH, Allers L, Salemi M, Phinney B, Johansen T, Deretic V (Abril de 2018). «Galectins Control mTOR in Response to Endomembrane Damage». Molecular Cell. 70 (1): 120–135.e8. PMC 5911935
 . PMID 29625033. doi:10.1016/j.molcel.2018.03.009
. PMID 29625033. doi:10.1016/j.molcel.2018.03.009
- ↑ Noda T, Ohsumi Y (Fevereiro de 1998). «Tor, a phosphatidylinositol kinase homologue, controls autophagy in yeast». The Journal of Biological Chemistry. 273 (7): 3963–6. PMID 9461583. doi:10.1074/jbc.273.7.3963

- ↑ Dubouloz F, Deloche O, Wanke V, Cameroni E, De Virgilio C (Julho de 2005). «The TOR and EGO protein complexes orchestrate microautophagy in yeast». Molecular Cell. 19 (1): 15–26. PMID 15989961. doi:10.1016/j.molcel.2005.05.020

- ↑ a b Ganley IG, Lam du H, Wang J, Ding X, Chen S, Jiang X (Maio de 2009). «ULK1.ATG13.FIP200 complex mediates mTOR signaling and is essential for autophagy». The Journal of Biological Chemistry. 284 (18): 12297–305. PMC 2673298
 . PMID 19258318. doi:10.1074/jbc.M900573200
. PMID 19258318. doi:10.1074/jbc.M900573200
- ↑ a b Jung CH, Jun CB, Ro SH, Kim YM, Otto NM, Cao J, Kundu M, Kim DH (Abril de 2009). «ULK-Atg13-FIP200 complexes mediate mTOR signaling to the autophagy machinery». Molecular Biology of the Cell. 20 (7): 1992–2003. PMC 2663920
 . PMID 19225151. doi:10.1091/mbc.e08-12-1249
. PMID 19225151. doi:10.1091/mbc.e08-12-1249
- ↑ a b Hosokawa N, Hara T, Kaizuka T, Kishi C, Takamura A, Miura Y, Iemura S, Natsume T, Takehana K, Yamada N, Guan JL, Oshiro N, Mizushima N (Abril de 2009). «Nutrient-dependent mTORC1 association with the ULK1-Atg13-FIP200 complex required for autophagy». Molecular Biology of the Cell. 20 (7): 1981–91. PMC 2663915
 . PMID 19211835. doi:10.1091/mbc.e08-12-1248
. PMID 19211835. doi:10.1091/mbc.e08-12-1248
- ↑ Hasegawa J, Maejima I, Iwamoto R, Yoshimori T (Março de 2015). «Selective autophagy: lysophagy». Methods. 75: 128–32. PMID 25542097. doi:10.1016/j.ymeth.2014.12.014

- ↑ Fraiberg M, Elazar Z (Outubro de 2016). «A TRIM16-Galactin3 Complex Mediates Autophagy of Damaged Endomembranes». Developmental Cell. 39 (1): 1–2. PMID 27728777. doi:10.1016/j.devcel.2016.09.025

- ↑ a b c Chauhan S, Kumar S, Jain A, Ponpuak M, Mudd MH, Kimura T, Choi SW, Peters R, Mandell M, Bruun JA, Johansen T, Deretic V (Outubro de 2016). «TRIMs and Galectins Globally Cooperate and TRIM16 and Galectin-3 Co-direct Autophagy in Endomembrane Damage Homeostasis». Developmental Cell. 39 (1): 13–27. PMC 5104201
 . PMID 27693506. doi:10.1016/j.devcel.2016.08.003
. PMID 27693506. doi:10.1016/j.devcel.2016.08.003
- ↑ Nishimura T, Kaizuka T, Cadwell K, Sahani MH, Saitoh T, Akira S, Virgin HW, Mizushima N (Março de 2013). «FIP200 regulates targeting of Atg16L1 to the isolation membrane». EMBO Reports. 14 (3): 284–91. PMC 3589088
 . PMID 23392225. doi:10.1038/embor.2013.6
. PMID 23392225. doi:10.1038/embor.2013.6
- ↑ Gammoh N, Florey O, Overholtzer M, Jiang X (Fevereiro de 2013). «Interaction between FIP200 and ATG16L1 distinguishes ULK1 complex-dependent and -independent autophagy». Nature Structural & Molecular Biology. 20 (2): 144–9. PMC 3565010
 . PMID 23262492. doi:10.1038/nsmb.2475
. PMID 23262492. doi:10.1038/nsmb.2475
- ↑ a b c Fujita N, Morita E, Itoh T, Tanaka A, Nakaoka M, Osada Y, Umemoto T, Saitoh T, Nakatogawa H, Kobayashi S, Haraguchi T, Guan JL, Iwai K, Tokunaga F, Saito K, Ishibashi K, Akira S, Fukuda M, Noda T, Yoshimori T (Outubro de 2013). «Recruitment of the autophagic machinery to endosomes during infection is mediated by ubiquitin». The Journal of Cell Biology. 203 (1): 115–28. PMC 3798248
 . PMID 24100292. doi:10.1083/jcb.201304188
. PMID 24100292. doi:10.1083/jcb.201304188
- ↑ Park JM, Jung CH, Seo M, Otto NM, Grunwald D, Kim KH, Moriarity B, Kim YM, Starker C, Nho RS, Voytas D, Kim DH (3 de março de 2016). «The ULK1 complex mediates MTORC1 signaling to the autophagy initiation machinery via binding and phosphorylating ATG14». Autophagy. 12 (3): 547–64. PMC 4835982
 . PMID 27046250. doi:10.1080/15548627.2016.1140293
. PMID 27046250. doi:10.1080/15548627.2016.1140293
- ↑ Dooley HC, Razi M, Polson HE, Girardin SE, Wilson MI, Tooze SA (Julho de 2014). «WIPI2 links LC3 conjugation with PI3P, autophagosome formation, and pathogen clearance by recruiting Atg12-5-16L1». Molecular Cell. 55 (2): 238–52. PMC 4104028
 . PMID 24954904. doi:10.1016/j.molcel.2014.05.021
. PMID 24954904. doi:10.1016/j.molcel.2014.05.021
- ↑ Kim J, Kundu M, Viollet B, Guan KL (Fevereiro de 2011). «AMPK and mTOR regulate autophagy through direct phosphorylation of Ulk1». Nature Cell Biology. 13 (2): 132–41. PMC 3987946
 . PMID 21258367. doi:10.1038/ncb2152
. PMID 21258367. doi:10.1038/ncb2152
- ↑ Kim J, Kim YC, Fang C, Russell RC, Kim JH, Fan W, Liu R, Zhong Q, Guan KL (Janeiro de 2013). «Differential regulation of distinct Vps34 complexes by AMPK in nutrient stress and autophagy». Cell. 152 (1–2): 290–303. PMC 3587159
 . PMID 23332761. doi:10.1016/j.cell.2012.12.016
. PMID 23332761. doi:10.1016/j.cell.2012.12.016
- ↑ Gwinn DM, Shackelford DB, Egan DF, Mihaylova MM, Mery A, Vasquez DS, Turk BE, Shaw RJ (Abril de 2008). «AMPK phosphorylation of raptor mediates a metabolic checkpoint». Molecular Cell. 30 (2): 214–26. PMC 2674027
 . PMID 18439900. doi:10.1016/j.molcel.2008.03.003
. PMID 18439900. doi:10.1016/j.molcel.2008.03.003
- ↑ Inoki K, Zhu T, Guan KL (Novembro de 2003). «TSC2 mediates cellular energy response to control cell growth and survival». Cell. 115 (5): 577–90. PMID 14651849. doi:10.1016/S0092-8674(03)00929-2

- ↑ Yoshida Y, Yasuda S, Fujita T, Hamasaki M, Murakami A, Kawawaki J, Iwai K, Saeki Y, Yoshimori T, Matsuda N, Tanaka K (Agosto de 2017). «FBXO27 directs damaged lysosomes for autophagy». Proceedings of the National Academy of Sciences of the United States of America. 114 (32): 8574–8579. PMC 5559013
 . PMID 28743755. doi:10.1073/pnas.1702615114
. PMID 28743755. doi:10.1073/pnas.1702615114
- ↑ Jimenez SA, Cronin PM, Koenig AS, O'Brien MS, Castro SV (15 Fevereiro de 2012). Varga J, Talavera F, Goldberg E, Mechaber AJ, Diamond HS, eds. «Scleroderma». Medscape Reference. WebMD. Consultado em 5 de março de 2014
- ↑ Hajj-ali RA (Junho de 2013). «Systemic Sclerosis». Merck Manual Professional. Merck Sharp & Dohme Corp. Consultado em 5 de março de 2014
- ↑ «Mammalian target of rapamycin (mTOR) inhibitors in solid tumours». Pharmaceutical Journal (em inglês). Consultado em 18 de outubro de 2018
- ↑ Faivre S, Kroemer G, Raymond E (Agosto de 2006). «Current development of mTOR inhibitors as anticancer agents». Nature Reviews. Drug Discovery (em inglês). 5 (8): 671–88. PMID 16883305. doi:10.1038/nrd2062
- ↑ Hasty P (Fevereiro de 2010). «Rapamycin: the cure for all that ails». Journal of Molecular Cell Biology. 2 (1): 17–9. PMID 19805415. doi:10.1093/jmcb/mjp033

- ↑ Bové J, Martínez-Vicente M, Vila M (Agosto de 2011). «Fighting neurodegeneration with rapamycin: mechanistic insights». Nature Reviews. Neuroscience. 12 (8): 437–52. PMID 21772323. doi:10.1038/nrn3068
- ↑ Mannick JB, Morris M, Hockey HP, Roma G, Beibel M, Kulmatycki K, Watkins M, Shavlakadze T, Zhou W, Quinn D, Glass DJ, Klickstein LB (Julho de 2018). «TORC1 inhibition enhances immune function and reduces infections in the elderly». Science Translational Medicine. 10 (449): eaaq1564. PMID 29997249. doi:10.1126/scitranslmed.aaq1564

- ↑ Estrela JM, Ortega A, Mena S, Rodriguez ML, Asensi M (2013). «Pterostilbene: Biomedical applications». Critical Reviews in Clinical Laboratory Sciences. 50 (3): 65–78. PMID 23808710. doi:10.3109/10408363.2013.805182
- ↑ McCubrey JA, Lertpiriyapong K, Steelman LS, Abrams SL, Yang LV, Murata RM, Rosalen PL, Scalisi A, Neri LM, Cocco L, Ratti S, Martelli AM, Laidler P, Dulińska-Litewka J, Rakus D, Gizak A, Lombardi P, Nicoletti F, Candido S, Libra M, Montalto G, Cervello M (Junho de 2017). «Effects of resveratrol, curcumin, berberine and other nutraceuticals on aging, cancer development, cancer stem cells and microRNAs». Aging. 9 (6): 1477–1536. PMC 5509453
 . PMID 28611316. doi:10.18632/aging.101250
. PMID 28611316. doi:10.18632/aging.101250
- ↑ Malavolta M, Bracci M, Santarelli L, Sayeed MA, Pierpaoli E, Giacconi R, Costarelli L, Piacenza F, Basso A, Cardelli M, Provinciali M (2018). «Inducers of Senescence, Toxic Compounds, and Senolytics: The Multiple Faces of Nrf2-Activating Phytochemicals in Cancer Adjuvant Therapy». Mediators of Inflammation. 2018. 4159013 páginas. PMC 5829354
 . PMID 29618945. doi:10.1155/2018/4159013
. PMID 29618945. doi:10.1155/2018/4159013
- ↑ Gómez-Linton DR, Alavez S, Alarcón-Aguilar A, López-Diazguerrero NE, Konigsberg M, Pérez-Flores LJ (Outubro de 2019). «Some naturally occurring compounds that increase longevity and stress resistance in model organisms of aging». Biogerontology. 20 (5): 583–603. PMID 31187283. doi:10.1007/s10522-019-09817-2
- ↑ Li W, Qin L, Feng R, Hu G, Sun H, He Y, Zhang R (Julho de 2019). «Emerging senolytic agents derived from natural products». Mechanisms of Ageing and Development. 181: 1–6. PMID 31077707. doi:10.1016/j.mad.2019.05.001
- ↑ «mTOR protein interactors». Human Protein Reference Database. Johns Hopkins University and the Institute of Bioinformatics. Consultado em 6 de dezembro de 2010. Cópia arquivada em 28 de junho de 2015
- ↑ Kumar V, Sabatini D, Pandey P, Gingras AC, Majumder PK, Kumar M, Yuan ZM, Carmichael G, Weichselbaum R, Sonenberg N, Kufe D, Kharbanda S (Abril de 2000). «Regulation of the rapamycin and FKBP-target 1/mammalian target of rapamycin and cap-dependent initiation of translation by the c-Abl protein-tyrosine kinase». The Journal of Biological Chemistry. 275 (15): 10779–87. PMID 10753870. doi:10.1074/jbc.275.15.10779

- ↑ Sekulić A, Hudson CC, Homme JL, Yin P, Otterness DM, Karnitz LM, Abraham RT (Julho de 2000). «A direct linkage between the phosphoinositide 3-kinase-AKT signaling pathway and the mammalian target of rapamycin in mitogen-stimulated and transformed cells». Cancer Research. 60 (13): 3504–13. PMID 10910062
- ↑ Cheng SW, Fryer LG, Carling D, Shepherd PR (Abril de 2004). «Thr2446 is a novel mammalian target of rapamycin (mTOR) phosphorylation site regulated by nutrient status». The Journal of Biological Chemistry. 279 (16): 15719–22. PMID 14970221. doi:10.1074/jbc.C300534200

- ↑ Choi JH, Bertram PG, Drenan R, Carvalho J, Zhou HH, Zheng XF (Outubro de 2002). «The FKBP12-rapamycin-associated protein (FRAP) is a CLIP-170 kinase». EMBO Reports. 3 (10): 988–94. PMC 1307618
 . PMID 12231510. doi:10.1093/embo-reports/kvf197
. PMID 12231510. doi:10.1093/embo-reports/kvf197
- ↑ Harris TE, Chi A, Shabanowitz J, Hunt DF, Rhoads RE, Lawrence JC (Abril de 2006). «mTOR-dependent stimulation of the association of eIF4G and eIF3 by insulin». The EMBO Journal. 25 (8): 1659–68. PMC 1440840
 . PMID 16541103. doi:10.1038/sj.emboj.7601047
. PMID 16541103. doi:10.1038/sj.emboj.7601047
- ↑ a b Schalm SS, Fingar DC, Sabatini DM, Blenis J (Maio de 2003). «TOS motif-mediated raptor binding regulates 4E-BP1 multisite phosphorylation and function». Current Biology. 13 (10): 797–806. Bibcode:2003CBio...13..797S. PMID 12747827. doi:10.1016/S0960-9822(03)00329-4

- ↑ a b c Hara K, Maruki Y, Long X, Yoshino K, Oshiro N, Hidayat S, Tokunaga C, Avruch J, Yonezawa K (Julho de 2002). «Raptor, a binding partner of target of rapamycin (TOR), mediates TOR action». Cell. 110 (2): 177–89. PMID 12150926. doi:10.1016/S0092-8674(02)00833-4

- ↑ a b Wang L, Rhodes CJ, Lawrence JC (Agosto de 2006). «Activation of mammalian target of rapamycin (mTOR) by insulin is associated with stimulation of 4EBP1 binding to dimeric mTOR complex 1». The Journal of Biological Chemistry. 281 (34): 24293–303. PMID 16798736. doi:10.1074/jbc.M603566200

- ↑ a b c Long X, Lin Y, Ortiz-Vega S, Yonezawa K, Avruch J (Abril de 2005). «Rheb binds and regulates the mTOR kinase». Current Biology. 15 (8): 702–13. Bibcode:2005CBio...15..702L. PMID 15854902. doi:10.1016/j.cub.2005.02.053

- ↑ a b Takahashi T, Hara K, Inoue H, Kawa Y, Tokunaga C, Hidayat S, Yoshino K, Kuroda Y, Yonezawa K (Setembro de 2000). «Carboxyl-terminal region conserved among phosphoinositide-kinase-related kinases is indispensable for mTOR function in vivo and in vitro». Genes to Cells. 5 (9): 765–75. PMID 10971657. doi:10.1046/j.1365-2443.2000.00365.x
- ↑ a b Burnett PE, Barrow RK, Cohen NA, Snyder SH, Sabatini DM (Fevereiro de 1998). «RAFT1 phosphorylation of the translational regulators p70 S6 kinase and 4E-BP1». Proceedings of the National Academy of Sciences of the United States of America. 95 (4): 1432–7. Bibcode:1998PNAS...95.1432B. PMC 19032
 . PMID 9465032. doi:10.1073/pnas.95.4.1432
. PMID 9465032. doi:10.1073/pnas.95.4.1432
- ↑ Wang X, Beugnet A, Murakami M, Yamanaka S, Proud CG (Abril de 2005). «Distinct signaling events downstream of mTOR cooperate to mediate the effects of amino acids and insulin on initiation factor 4E-binding proteins». Molecular and Cellular Biology. 25 (7): 2558–72. PMC 1061630
 . PMID 15767663. doi:10.1128/MCB.25.7.2558-2572.2005
. PMID 15767663. doi:10.1128/MCB.25.7.2558-2572.2005
- ↑ a b c Jacinto E, Loewith R, Schmidt A, Lin S, Rüegg MA, Hall A, Hall MN (Novembro de 2004). «Mammalian TOR complex 2 controls the actin cytoskeleton and is rapamycin insensitive». Nature Cell Biology. 6 (11): 1122–8. PMID 15467718. doi:10.1038/ncb1183
- ↑ Choi J, Chen J, Schreiber SL, Clardy J (Julho de 1996). «Structure of the FKBP12-rapamycin complex interacting with the binding domain of human FRAP». Science. 273 (5272): 239–42. Bibcode:1996Sci...273..239C. PMID 8662507. doi:10.1126/science.273.5272.239
- ↑ Luker KE, Smith MC, Luker GD, Gammon ST, Piwnica-Worms H, Piwnica-Worms D (Agosto de 2004). «Kinetics of regulated protein-protein interactions revealed with firefly luciferase complementation imaging in cells and living animals». Proceedings of the National Academy of Sciences of the United States of America. 101 (33): 12288–93. Bibcode:2004PNAS..10112288L. PMC 514471
 . PMID 15284440. doi:10.1073/pnas.0404041101
. PMID 15284440. doi:10.1073/pnas.0404041101
- ↑ Banaszynski LA, Liu CW, Wandless TJ (Abril de 2005). «Characterization of the FKBP.rapamycin.FRB ternary complex». Journal of the American Chemical Society. 127 (13): 4715–21. PMID 15796538. doi:10.1021/ja043277y
- ↑ Sabers CJ, Martin MM, Brunn GJ, Williams JM, Dumont FJ, Wiederrecht G, Abraham RT (Janeiro de 1995). «Isolation of a protein target of the FKBP12-rapamycin complex in mammalian cells». The Journal of Biological Chemistry. 270 (2): 815–22. PMID 7822316. doi:10.1074/jbc.270.2.815

- ↑ Sabatini DM, Barrow RK, Blackshaw S, Burnett PE, Lai MM, Field ME, Bahr BA, Kirsch J, Betz H, Snyder SH (Maio de 1999). «Interaction of RAFT1 with gephyrin required for rapamycin-sensitive signaling». Science. 284 (5417): 1161–4. Bibcode:1999Sci...284.1161S. PMID 10325225. doi:10.1126/science.284.5417.1161
- ↑ Ha SH, Kim DH, Kim IS, Kim JH, Lee MN, Lee HJ, Kim JH, Jang SK, Suh PG, Ryu SH (Dezembro de 2006). «PLD2 forms a functional complex with mTOR/raptor to transduce mitogenic signals». Cellular Signalling. 18 (12): 2283–91. PMID 16837165. doi:10.1016/j.cellsig.2006.05.021
- ↑ Buerger C, DeVries B, Stambolic V (Junho de 2006). «Localization of Rheb to the endomembrane is critical for its signaling function». Biochemical and Biophysical Research Communications. 344 (3): 869–80. PMID 16631613. doi:10.1016/j.bbrc.2006.03.220
- ↑ a b Jacinto E, Facchinetti V, Liu D, Soto N, Wei S, Jung SY, Huang Q, Qin J, Su B (Outubro de 2006). «SIN1/MIP1 maintains rictor-mTOR complex integrity and regulates Akt phosphorylation and substrate specificity». Cell. 127 (1): 125–37. PMID 16962653. doi:10.1016/j.cell.2006.08.033

- ↑ McMahon LP, Yue W, Santen RJ, Lawrence JC (Janeiro de 2005). «Farnesylthiosalicylic acid inhibits mammalian target of rapamycin (mTOR) activity both in cells and in vitro by promoting dissociation of the mTOR-raptor complex». Molecular Endocrinology. 19 (1): 175–83. PMID 15459249. doi:10.1210/me.2004-0305

- ↑ Oshiro N, Yoshino K, Hidayat S, Tokunaga C, Hara K, Eguchi S, Avruch J, Yonezawa K (Abril de 2004). «Dissociation of raptor from mTOR is a mechanism of rapamycin-induced inhibition of mTOR function». Genes to Cells. 9 (4): 359–66. PMID 15066126. doi:10.1111/j.1356-9597.2004.00727.x. hdl:20.500.14094/D1002969

- ↑ Kawai S, Enzan H, Hayashi Y, Jin YL, Guo LM, Miyazaki E, Toi M, Kuroda N, Hiroi M, Saibara T, Nakayama H (Julho de 2003). «Vinculin: a novel marker for quiescent and activated hepatic stellate cells in human and rat livers». Virchows Archiv. 443 (1): 78–86. PMID 12719976. doi:10.1007/s00428-003-0804-4
- ↑ Choi KM, McMahon LP, Lawrence JC (Maio de 2003). «Two motifs in the translational repressor PHAS-I required for efficient phosphorylation by mammalian target of rapamycin and for recognition by raptor». The Journal of Biological Chemistry. 278 (22): 19667–73. PMID 12665511. doi:10.1074/jbc.M301142200

- ↑ a b Nojima H, Tokunaga C, Eguchi S, Oshiro N, Hidayat S, Yoshino K, Hara K, Tanaka N, Avruch J, Yonezawa K (Maio de 2003). «The mammalian target of rapamycin (mTOR) partner, raptor, binds the mTOR substrates p70 S6 kinase and 4E-BP1 through their TOR signaling (TOS) motif». The Journal of Biological Chemistry. 278 (18): 15461–4. PMID 12604610. doi:10.1074/jbc.C200665200

- ↑ a b Sarbassov DD, Ali SM, Sengupta S, Sheen JH, Hsu PP, Bagley AF, Markhard AL, Sabatini DM (Abril de 2006). «Prolonged rapamycin treatment inhibits mTORC2 assembly and Akt/PKB». Molecular Cell. 22 (2): 159–68. PMID 16603397. doi:10.1016/j.molcel.2006.03.029

- ↑ Tzatsos A, Kandror KV (Janeiro de 2006). «Nutrients suppress phosphatidylinositol 3-kinase/Akt signaling via raptor-dependent mTOR-mediated insulin receptor substrate 1 phosphorylation». Molecular and Cellular Biology. 26 (1): 63–76. PMC 1317643
 . PMID 16354680. doi:10.1128/MCB.26.1.63-76.2006
. PMID 16354680. doi:10.1128/MCB.26.1.63-76.2006
- ↑ a b c Sarbassov DD, Sabatini DM (Novembro de 2005). «Redox regulation of the nutrient-sensitive raptor-mTOR pathway and complex». The Journal of Biological Chemistry. 280 (47): 39505–9. PMID 16183647. doi:10.1074/jbc.M506096200

- ↑ a b Yang Q, Inoki K, Ikenoue T, Guan KL (Outubro de 2006). «Identification of Sin1 as an essential TORC2 component required for complex formation and kinase activity». Genes & Development. 20 (20): 2820–32. PMC 1619946
 . PMID 17043309. doi:10.1101/gad.1461206
. PMID 17043309. doi:10.1101/gad.1461206
- ↑ Kumar V, Pandey P, Sabatini D, Kumar M, Majumder PK, Bharti A, Carmichael G, Kufe D, Kharbanda S (Março de 2000). «Functional interaction between RAFT1/FRAP/mTOR and protein kinase cdelta in the regulation of cap-dependent initiation of translation». The EMBO Journal. 19 (5): 1087–97. PMC 305647
 . PMID 10698949. doi:10.1093/emboj/19.5.1087
. PMID 10698949. doi:10.1093/emboj/19.5.1087
- ↑ Long X, Ortiz-Vega S, Lin Y, Avruch J (Junho de 2005). «Rheb binding to mammalian target of rapamycin (mTOR) is regulated by amino acid sufficiency». The Journal of Biological Chemistry. 280 (25): 23433–6. PMID 15878852. doi:10.1074/jbc.C500169200

- ↑ Smith EM, Finn SG, Tee AR, Browne GJ, Proud CG (Maio de 2005). «The tuberous sclerosis protein TSC2 is not required for the regulation of the mammalian target of rapamycin by amino acids and certain cellular stresses». The Journal of Biological Chemistry. 280 (19): 18717–27. PMID 15772076. doi:10.1074/jbc.M414499200

- ↑ Bernardi R, Guernah I, Jin D, Grisendi S, Alimonti A, Teruya-Feldstein J, Cordon-Cardo C, Simon MC, Rafii S, Pandolfi PP (Agosto de 2006). «PML inhibits HIF-1alpha translation and neoangiogenesis through repression of mTOR». Nature. 442 (7104): 779–85. Bibcode:2006Natur.442..779B. PMID 16915281. doi:10.1038/nature05029
- ↑ Saitoh M, Pullen N, Brennan P, Cantrell D, Dennis PB, Thomas G (Maio de 2002). «Regulation of an activated S6 kinase 1 variant reveals a novel mammalian target of rapamycin phosphorylation site». The Journal of Biological Chemistry. 277 (22): 20104–12. PMID 11914378. doi:10.1074/jbc.M201745200

- ↑ Chiang GG, Abraham RT (Julho de 2005). «Phosphorylation of mammalian target of rapamycin (mTOR) at Ser-2448 is mediated by p70S6 kinase». The Journal of Biological Chemistry. 280 (27): 25485–90. PMID 15899889. doi:10.1074/jbc.M501707200

- ↑ Holz MK, Blenis J (Julho de 2005). «Identification of S6 kinase 1 as a novel mammalian target of rapamycin (mTOR)-phosphorylating kinase». The Journal of Biological Chemistry. 280 (28): 26089–93. PMID 15905173. doi:10.1074/jbc.M504045200

- ↑ Isotani S, Hara K, Tokunaga C, Inoue H, Avruch J, Yonezawa K (Novembro de 1999). «Immunopurified mammalian target of rapamycin phosphorylates and activates p70 S6 kinase alpha in vitro». The Journal of Biological Chemistry. 274 (48): 34493–8. PMID 10567431. doi:10.1074/jbc.274.48.34493
 . hdl:20.500.14094/D1002182
. hdl:20.500.14094/D1002182
- ↑ Toral-Barza L, Zhang WG, Lamison C, Larocque J, Gibbons J, Yu K (Junho de 2005). «Characterization of the cloned full-length and a truncated human target of rapamycin: activity, specificity, and enzyme inhibition as studied by a high capacity assay». Biochemical and Biophysical Research Communications. 332 (1): 304–10. PMID 15896331. doi:10.1016/j.bbrc.2005.04.117
- ↑ Ali SM, Sabatini DM (Maio de 2005). «Structure of S6 kinase 1 determines whether raptor-mTOR or rictor-mTOR phosphorylates its hydrophobic motif site». The Journal of Biological Chemistry. 280 (20): 19445–8. PMID 15809305. doi:10.1074/jbc.C500125200

- ↑ Edinger AL, Linardic CM, Chiang GG, Thompson CB, Abraham RT (Dezembro de 2003). «Differential effects of rapamycin on mammalian target of rapamycin signaling functions in mammalian cells». Cancer Research. 63 (23): 8451–60. PMID 14679009
- ↑ Leone M, Crowell KJ, Chen J, Jung D, Chiang GG, Sareth S, Abraham RT, Pellecchia M (Agosto de 2006). «The FRB domain of mTOR: NMR solution structure and inhibitor design». Biochemistry. 45 (34): 10294–302. PMID 16922504. doi:10.1021/bi060976+
- ↑ Kristof AS, Marks-Konczalik J, Billings E, Moss J (Setembro de 2003). «Stimulation of signal transducer and activator of transcription-1 (STAT1)-dependent gene transcription by lipopolysaccharide and interferon-gamma is regulated by mammalian target of rapamycin». The Journal of Biological Chemistry. 278 (36): 33637–44. PMID 12807916. doi:10.1074/jbc.M301053200

- ↑ Yokogami K, Wakisaka S, Avruch J, Reeves SA (Janeiro de 2000). «Serine phosphorylation and maximal activation of STAT3 during CNTF signaling is mediated by the rapamycin target mTOR». Current Biology. 10 (1): 47–50. Bibcode:2000CBio...10...47Y. PMID 10660304. doi:10.1016/S0960-9822(99)00268-7

- ↑ Kusaba H, Ghosh P, Derin R, Buchholz M, Sasaki C, Madara K, Longo DL (Janeiro de 2005). «Interleukin-12-induced interferon-gamma production by human peripheral blood T cells is regulated by mammalian target of rapamycin (mTOR)». The Journal of Biological Chemistry. 280 (2): 1037–43. PMID 15522880. doi:10.1074/jbc.M405204200

- ↑ Cang C, Zhou Y, Navarro B, Seo YJ, Aranda K, Shi L, Battaglia-Hsu S, Nissim I, Clapham DE, Ren D (Fevereiro de 2013). «mTOR regulates lysosomal ATP-sensitive two-pore Na(+) channels to adapt to metabolic state». Cell. 152 (4): 778–90. PMC 3908667
 . PMID 23394946. doi:10.1016/j.cell.2013.01.023
. PMID 23394946. doi:10.1016/j.cell.2013.01.023
- ↑ Wu S, Mikhailov A, Kallo-Hosein H, Hara K, Yonezawa K, Avruch J (Janeiro de 2002). «Characterization of ubiquilin 1, an mTOR-interacting protein». Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1542 (1–3): 41–56. PMID 11853878. doi:10.1016/S0167-4889(01)00164-1

Leitura adicional
[editar | editar código-fonte]- Saxton RA, Sabatini DM (Março de 2017). «mTOR Signaling in Growth, Metabolism, and Disease». Cell. 168 (6): 960–976. PMC 5394987
 . PMID 28283069. doi:10.1016/j.cell.2017.02.004
. PMID 28283069. doi:10.1016/j.cell.2017.02.004
Ligações externas
[editar | editar código-fonte]- MeSH mTOR+protein
- «mTOR Signaling Pathway in Pathway Interaction Database». National Cancer Institute. Consultado em 18 de outubro de 2015. Cópia arquivada em 18 de março de 2013
- Visão geral de todas as informações estruturais disponíveis no PDB para UniProt: P42345 (Serina/treonina-proteína quinase mTOR) no PDBe-KB.