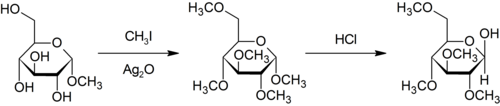Metylering
Metylering inom de kemiska vetenskaperna, är tillägg av en metylgrupp på ett substrat, eller utbyte av en atom (eller grupp) med en metylgrupp. Metylering är en form av alkylering, med en metylgrupp som ersätter en väteatom. Dessa termer används ofta inom kemi, biokemi, markvetenskap och biologi.
I biologiska system katalyseras metylering av enzymer. Sådan metylering kan ske vid modifiering av tungmetaller, reglering av genuttryck, reglering av proteinfunktion och RNA-bearbetning. In vitro-metylering av vävnadsprover är också ett sätt att minska vissa histologiska färgningsartefakter. Motsatsen till metylering är demetylering.
Inom biologi
[redigera | redigera wikitext]I biologiska system åstadkoms metylering av enzymer. Metylering kan modifiera tungmetaller och kan reglera genuttryck, RNA-bearbetning och proteinfunktion. Det är en nyckelprocess som ligger bakom epigenetik.
Biometanogenes
[redigera | redigera wikitext]Metanogenes, processen som genererar metan från CO2, involverar en serie metyleringsreaktioner. Dessa reaktioner orsakas av en uppsättning enzymer som härbärgeras av en familj av anaeroba mikrober.[1]

I omvänd metanogenes är metan metyleringsmedlet
O-metyltransferaser
[redigera | redigera wikitext]En mängd olika fenoler genomgår O-metylering för att ge anisolderivat. Denna process, katalyserad av sådana enzymer som caffeoyl-CoA O-metyltransferas, är en nyckelreaktion i biosyntesen av lignoler, föregångare till lignin, en viktig strukturell komponent i växter.
Växter producerar flavonoider och isoflavoner med metyleringar på hydroxylgrupper, det vill säga metoxibindningar. Denna 5-O-metylering påverkar flavonoidens vattenlöslighet. Exempel är 5-O-metylgenistein, 5-O-metylmyricetin och 5-O-metylquercetin (azaleatin).
Proteiner
[redigera | redigera wikitext]Tillsammans med ubiquitinering och fosforylering är metylering en viktig biokemisk process för att modifiera proteinfunktionen. De vanligaste proteinmetyleringarna påverkar arginin och lysinrester av specifika histoner. Annars är histidin, glutamat, asparagin, cystein mottagliga för metylering. Några av dessa produkter inkluderar S-metylcystein, två isomerer av N-metylhistidin och två isomerer av N-metylarginin.[2]
Metioninsyntas
[redigera | redigera wikitext]
Metioninsyntas regenererar metionin (Met) från homocystein (Hcy). Den totala reaktionen omvandlar 5-metyltetrahydrofolat (N5-MeTHF) till tetrahydrofolat (THF) samtidigt som en metylgrupp överförs till Hcy för att bilda Met. Metioninsynteser kan vara kobalaminberoende och kobalaminoberoende: Växter har båda, djur är beroende av den metylkobalaminberoende formen.
I metylkobalaminberoende former av enzymet fortskrider reaktionen i två steg i en ping-pong-reaktion. Enzymet förbereds initialt till ett reaktivt tillstånd genom överföring av en metylgrupp från N5-MeTHF till Co(I) i enzymbundet kobalamin (Cob), vilket bildar metyl-kobalamin (Me-Cob) som nu innehåller Me-Co( III) och aktiverar enzymet. Sedan reagerar en Hcy som har koordinerats till en enzymbunden zink för att bilda ett reaktivt tiolat med Me-Cob. Den aktiverade metylgruppen överförs från Me-Cob till Hcy-tiolatet, som regenererar Co(I) i Cob, och Met frigörs från enzymet.[3]
Tungmetaller: arsenik, kvicksilver, kadmium
[redigera | redigera wikitext]Biometylering är vägen för att omvandla vissa tunga grundämnen till mer rörliga eller mer dödliga derivat som kan komma in i näringskedjan. Biometyleringen av arsenikföreningar börjar med bildandet av metanarsonater. Således metyleras trevärda oorganiska arsenikföreningar för att ge metanearsonat. S-adenosylmetionin är metyldonatorn. Metanarsonaten är råvara till dimetylarsonater, återigen genom reduktionscykeln (till metylarsonsyra) följt av en andra metylering.[4] Besläktade vägar finns i mikrobiell metylering av kvicksilver till metylkvicksilver.
Epigenetisk metylering
[redigera | redigera wikitext]DNA/RNA-metylering
[redigera | redigera wikitext]DNA-metylering är omvandlingen av cytosinet till 5-metylcytosin. Bildandet av Me-CpG katalyseras av enzymet DNA-metyltransferas. Hos ryggradsdjur sker DNA-metylering vanligtvis vid CpG-ställen (cytosin-fosfat-guanin-ställen - det vill säga platser där ett cytosin direkt följs av ett guanin i DNA-sekvensen). Hos däggdjur är DNA-metylering vanlig i kroppsceller,[5] och metylering av CpG-ställen verkar vara standard.[6][7] Humant DNA har cirka 80–90 procent av CpG-ställena metylerade, men det finns vissa områden, kända som CpG-öar, som är CG-rika (högt cytosin- och guanininnehåll, som består av cirka 65 procent CG-rester), där ingen är metylerad . Dessa är förbundna med promotorerna för 56 procent av däggdjursgener, inklusive alla allmänt uttryckta gener. En till två procent av det mänskliga genomet är CpG-kluster, och det finns ett omvänt samband mellan CpG-metylering och transkriptionsaktivitet. Metylering som bidrar till epigenetisk nedärvning kan ske genom antingen DNA-metylering eller proteinmetylering. Felaktig metylering av mänskliga gener kan leda till sjukdomsutveckling,[8][9] inklusive cancer.[10][11]
Hos honungsbin är DNA-metylering associerad med alternativ splitsning och genreglering enligt funktionell genomforskning publicerad 2013.[12] Dessutom är DNA-metylering kopplad till uttrycksförändringar i immungener när honungsbin är under dödlig virusinfektion.[13] Flera granskningsartiklar har publicerats om DNA-metylering hos sociala insekter.[14][15]
RNA-metylering förekommer i olika RNA-arter, det vill säga tRNA, rRNA, mRNA, tmRNA, snRNA, snoRNA, miRNA och viralt RNA. Olika katalytiska strategier används för RNA-metylering av en mängd olika RNA-metyltransferaser. RNA-metylering tros ha funnits innan DNA-metylering i de tidiga livsformerna som utvecklades på jorden.[16]
N6-metyladenosin (m6A) är den vanligaste förekommande metyleringsmodifieringen i RNA-molekyler (mRNA) som finns i eukaryoter. 5-metylcytosin (5-mC) förekommer också vanligtvis i olika RNA-molekyler. Nya data tyder starkt på att m6A- och 5-mC RNA-metylering påverkar regleringen av olika biologiska processer såsom RNA-stabilitet och mRNA-translation,[17] och att onormal RNA-metylering bidrar till etiologin för mänskliga sjukdomar.[18]
Hos sociala insekter som honungsbin studeras RNA-metylering som en möjlig epigenetisk mekanism som ligger bakom aggression via ömsesidiga korsningar.[19]
Proteinmetylering
[redigera | redigera wikitext]Proteinmetylering sker vanligtvis på arginin- eller lysinaminosyrarester i proteinsekvensen.[20] Arginin kan metyleras en gång (monometylerat arginin) eller två gånger, med antingen båda metylgrupperna på ett terminalt kväve (asymmetriskt dimetylarginin) eller en på båda kväveatomerna (symmetriskt dimetylarginin), av proteinargininmetyltransferaser (PRMT). Lysin kan metyleras en, två eller tre gånger med lysinmetyltransferaser. Proteinmetylering har studerats mest i histoner. Överföringen av metylgrupper från S-adenosylmetionin till histoner katalyseras av enzymer som kallas histonmetyltransferaser. Histoner som är metylerade på vissa rester kan verka epigenetiskt för att undertrycka eller aktivera genuttryck.[21][22]Proteinmetylering är en typ av posttranslationell modifiering.
Utveckling
[redigera | redigera wikitext]Metylmetabolism är mycket gammal och kan hittas i alla organismer på jorden, från bakterier till människor, vilket visar vikten av metylmetabolism för fysiologin.[23] Faktum är att farmakologisk hämning av global metylering hos arter som sträcker sig från människa, mus, fisk, fluga, spolmask, växt, alger och cyanobakterier orsakar samma effekter på deras biologiska rytmer, vilket visar bevarade fysiologiska roller för metylering under evolutionen.[24]
Inom kemin
[redigera | redigera wikitext]Termen metylering i organisk kemi hänvisar till alkyleringsprocessen som används för att beskriva leveransen av en CH3-grupp.[25]
Elektrofil metylering
[redigera | redigera wikitext]Metyleringar utförs vanligtvis med användning av elektrofila metylkällor såsom jodmetan,[26] dimetylsulfat,[27][28] dimetylkarbonat[29] eller tetrametylammoniumklorid.[30]Mindre vanliga men mer kraftfulla (och farligare) metyleringsreagens är metyltriflat,[31] diazometan,[32] och metylfluorsulfonat (magisk metyl). Dessa reagens reagerar alla via SN2-nukleofila substitutioner. Till exempel kan ett karboxylat metyleras på syre för att ge en metylester, ett alkoxidsalt RO− kan likaledes metyleras för att ge en eter, ROCH3, eller ett ketonenolat kan metyleras på kol för att producera en ny keton.
Purdie-metyleringen är en specifik för metylering vid syre av kolhydrater med jodmetan och silveroxid.[33]
Eschweiler–Clarke-metylering
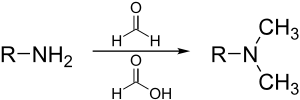
[redigera | redigera wikitext]Eschweiler-Clarke-reaktionen är en metod för metylering av aminer.[34] Denna metod undviker risken för kvaternisering, som uppstår när aminer metyleras med metylhalogenider.
Diazometan och trimetylsilyldiazometan
[redigera | redigera wikitext]Diazometan och den säkrare analogen trimetylsilyldiazometanmetylatkarboxylsyror, fenoler och till och med alkoholer:
Metoden ger fördelen att biprodukterna lätt tas bort från produktblandningen.[35]
Nukleofil metylering
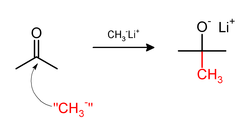
[redigera | redigera wikitext]Metylering involverar ibland användning av nukleofila metylreagens. Starkt nukleofila metyleringsmedel inkluderar metyllitium (CH3Li)[36] eller Grignard-reagenser som metylmagnesiumbromid (CH3MgX).[37] Till exempel kommer CH3Li att lägga till metylgrupper till karbonyl (C=O) av ketoner och aldehyd.
Mildare metyleringsmedel är tetrametyltenn, dimetylzink och trimetylaluminium.[38]
Se även
[redigera | redigera wikitext]Referenser
[redigera | redigera wikitext]- Den här artikeln är helt eller delvis baserad på material från engelskspråkiga Wikipedia, Methylation, 27 november 2023.
Noter
[redigera | redigera wikitext]- ^ Thauer, R. K., "Biochemistry of Methanogenesis: a Tribute to Marjory Stephenson", Microbiology, 1998, volume 144, pages 2377-2406.
- ^ Clarke, Steven G. (2018). ”The ribosome: A hot spot for the identification of new types of protein methyltransferases”. Journal of Biological Chemistry 293 (27): sid. 10438–10446. doi:. PMID 29743234.
- ^ Matthews, R. G.; Smith, A. E.; Zhou, Z. S.; Taurog, R. E.; Bandarian, V.; Evans, J. C.; Ludwig, M. (2003). ”Cobalamin-Dependent and Cobalamin-Independent Methionine Synthases: Are There Two Solutions to the Same Chemical Problem?”. Helvetica Chimica Acta 86 (12): sid. 3939–3954. doi:.
- ^ Styblo, M.; Del Razo, L. M.; Vega, L.; Germolec, D. R.; LeCluyse, E. L.; Hamilton, G. A.; Reed, W.; Wang, C.; et al. (2000). ”Comparative toxicity of trivalent and pentavalent inorganic and methylated arsenicals in rat and human cells”. Archives of Toxicology 74 (6): sid. 289–299. doi:. PMID 11005674.
- ^ Tost J (2010). ”DNA methylation: an introduction to the biology and the disease-associated changes of a promising biomarker.”. Mol Biotechnol 44 (1): sid. 71–81. doi:. PMID 19842073.
- ^ ”Human DNA methylomes at base resolution show widespread epigenomic differences”. Nature 462 (7271): sid. 315–22. November 2009. doi:. PMID 19829295. Bibcode: 2009Natur.462..315L.
- ^ ”DNA-binding factors shape the mouse methylome at distal regulatory regions”. Nature 480 (7378): sid. 490–5. December 2011. doi:. PMID 22170606.
- ^ ”Methylation loss at H19 imprinted gene correlates with methylenetetrahydrofolate reductase gene promoter hypermethylation in semen samples from infertile males”. Epigenetics 8 (9): sid. 990–7. September 2013. doi:. PMID 23975186.
- ^ ”Methylenetetrahydrofolate reductase gene promoter hypermethylation in semen samples of infertile couples correlates with recurrent spontaneous abortion”. Human Reproduction 27 (12): sid. 3632–8. December 2012. doi:. PMID 23010533. https://academic.oup.com/humrep/article/27/12/3632/651064.
- ^ ”Hypermethylation-Induced Inactivation of the IRF6 Gene as a Possible Early Event in Progression of Vulvar Squamous Cell Carcinoma Associated With Lichen Sclerosus”. JAMA Dermatology 152 (8): sid. 928–33. 2016. doi:. PMID 27223861.
- ^ ”Association of Retinoic Acid Receptor β Gene With Onset and Progression of Lichen Sclerosus-Associated Vulvar Squamous Cell Carcinoma”. JAMA Dermatology 154 (7): sid. 819–823. 2018. doi:. PMID 29898214.
- ^ Li-Byarlay et al., "RNA interference knockdown of DNA methyl-transferase 3 affects gene alternative splicing in the honey bee", Proceedings of the National Academy of Sciences 110 (31), 12750-12755, https://doi.org/10.1073/pnas.1310735110
- ^ Li-Byarlay et al. 2020, "Transcriptomic and epigenomic dynamics of honey bees in response to lethal viral infection" Frontiers in genetics 11, 1056, https://doi.org/10.3389/fgene.2020.566320
- ^ Li-Byarlay, "The Function of DNA Methylation Marks in Social Insects" Front. Ecol. Evol., 19 May 2016 Sec. Social Evolution, Volume 4 - 2016 https://doi.org/10.3389/fevo.2016.00057
- ^ Wang & Li-Byarlay, "Chapter Two-Physiological and Molecular Mechanisms of Nutrition in Honey Bees", 2015, Advances in Insect Physiology, 49: 25-58. https://www.sciencedirect.com/science/article/abs/pii/S0065280615000259
- ^ Rana, Ajay K.; Ankri, Serge (2016-01-01). ”Reviving the RNA World: An Insight into the Appearance of RNA Methyltransferases”. Front Genet 7: sid. 99. doi:. PMID 27375676.
- ^ Choi, Junhong; Ieong, Ka-Weng; Demirci, Hasan; Chen, Jin; Petrov, Alexey; Prabhakar, Arjun; O'Leary, Seán E.; Dominissini, Dan; et al. (February 2016). ”N6-methyladenosine in mRNA disrupts tRNA selection and translation-elongation dynamics” (på engelska). Nature Structural & Molecular Biology 23 (2): sid. 110–115. doi:. ISSN 1545-9993. PMID 26751643.
- ^ Stewart, Kendal (15 September 2017). ”Methylation (MTHFR) Testing & Folate Deficiency”. Methylation (MTHFR) Testing & Folate Deficiency. https://www.genomixnutrition.com/methylation-testing-s/2.htm.
- ^ Bresnahan et al, "Examining parent-of-origin effects on transcription and RNA methylation in mediating aggressive behavior in honey bees (Apis mellifera)" BMC Genomics volume 24, Article number: 315 (2023), https://doi.org/10.1186/s12864-023-09411-4
- ^ Walsh, Christopher (2006). ”Chapter 5 – Protein Methylation”. Posttranslational modification of proteins: expanding nature's inventory. Roberts and Co. Publishers. ISBN 978-0-9747077-3-0.[död länk]
- ^ Grewal, S. I.; Rice, J. C. (2004). ”Regulation of heterochromatin by histone methylation and small RNAs”. Current Opinion in Cell Biology 16 (3): sid. 230–238. doi:. PMID 15145346. https://zenodo.org/record/1258832.
- ^ Nakayama, J. -I.; Rice, J. C.; Strahl, B. D.; Allis, C. D.; Grewal, S. I. (2001). ”Role of Histone H3 Lysine 9 Methylation in Epigenetic Control of Heterochromatin Assembly”. Science 292 (5514): sid. 110–113. doi:. PMID 11283354. Bibcode: 2001Sci...292..110N.
- ^ Kozbial, P.Z.; Mushegian, A.R. (2005). ”Natural history of S-adenosylmethionine-binding proteins.”. BMC Struct Biol 5 (19): sid. 19. doi:. PMID 16225687.
- ^ Fustin, J.M.; Ye, S.; Rakers, C.; Kaneko, K.; Fukumoto, K.; Yamano, M.; Versteven, M.; Grünewald, E.; et al. (2020). ”Methylation deficiency disrupts biological rhythms from bacteria to humans.”. Communications Biology 3 (211): sid. 211. doi:. PMID 32376902.
- ^ March, Jerry; Smith, Michael W (2001). March's advanced organic chemistry: reactions, mechanisms, and structure. New York: Wiley. ISBN 978-0-471-58589-3.
- ^ Vyas, G. N.; Shah, N. M. (1951). ”Quninacetophenone monomethyl ether”. Organic Syntheses 31: sid. 90. doi:.
- ^ Hiers, G. S. (1929). ”Anisole”. Organic Syntheses 9: sid. 12. doi:.
- ^ Icke, Roland N.; Redemann, Ernst; Wisegarver, Burnett B.; Alles, Gordon A. (1949). ”m-Methoxybenzaldehyde”. Organic Syntheses 29: sid. 63. doi:.
- ^ Tundo, Pietro; Selva, Maurizio; Bomben, Andrea (1999). ”Mono-C-methylathion of arylacetonitriles and methyl arylacetates by dimethyl carbonate: a general method for the synthesis of pure 2-arylpropionic acids. 2-Phenylpropionic acid”. Organic Syntheses 76: sid. 169. doi:.
- ^ Nenad, Maraš; Polanc, Slovenko; Kočevar, Marijan (2008). ”Microwave-assisted methylation of phenols with tetramethylammonium chloride in the presence of K2CO3 or Cs2CO3”. Tetrahedron 64 (51): sid. 11618–11624. doi:.
- ^ Poon, Kevin W. C.; Albiniak, Philip A.; Dudley, Gregory B. (2007). ”Protection of alcohols using 2-benzyloxy-1-methylpyridinium trifluoromethanesulfanonate: Methyl (R)-(-)-3-benzyloxy-2-methyl propanoate”. Organic Syntheses 84: sid. 295. doi:.
- ^ Neeman, M.; Johnson, William S. (1961). ”Cholestanyl methyl ether”. Organic Syntheses 41: sid. 9. doi:.
- ^ Purdie, T.; Irvine, J. C. (1903). ”C.?The alkylation of sugars”. Journal of the Chemical Society, Transactions 83: sid. 1021–1037. doi:. https://zenodo.org/record/2039403.
- ^ Icke, Roland N.; Wisegarver, Burnett B.; Alles, Gordon A. (1945). ”β-Phenylethyldimethylamine”. Organic Syntheses 25: sid. 89. doi:.
- ^ ”Encyclopedia of Reagents for Organic Synthesis”. e-EROS Encyclopedia of Reagents for Organic Synthesis. 2001. doi:. ISBN 978-0-471-93623-7.
- ^ Lipsky, Sharon D.; Hall, Stan S. (1976). ”Aromatic Hydrocarbons from aromatic ketones and aldehydes: 1,1-Diphenylethane”. Organic Syntheses 55: sid. 7. doi:.
- ^ Grummitt, Oliver; Becker, Ernest I. (1950). ”trans-1-Phenyl-1,3-butadiene”. Organic Syntheses 30: sid. 75. doi:.
- ^ Negishi, Ei-ichi; Matsushita, Hajime (1984). ”Palladium-Catalyzed Synthesis of 1,4-Dienes by Allylation of Alkenyalane: α-Farnesene”. Organic Syntheses 62: sid. 31. doi:.
Externa länkar
[redigera | redigera wikitext] Wikimedia Commons har media som rör Metylering.
Wikimedia Commons har media som rör Metylering.
|